Guía de campo de R
PROLOGO
Con esta guía buscamos aportar los fundamentos básicos para utilizar R de la manera más eficiente. R es un programa con grandes capacidades, sin embargo, en esta guía nos enfocamos en el manejo de bases de datos y análisis básicos. En particular, como somos biólogos esta guía está sesgada al contexto de análisis de datos biológicos seguiendo la lógica de una guía de campo la cual, idealmente, debe ser concisa, liviana y bien ilustrada.
Para aquellos que comienzan a usar R, el manejo de bases de datos, modificar columnas y/o filas o la obtención de estadísticas básicas son uno de los principales retos. Muchas veces los principiantes se ven tentados a regresar a los viejos programas y dejan R para el momento de hacer análisis "sofisticados". Esta práctica es en realidad muy poco eficiente y cortoplacista ya que obtienes lo necesario en el momento pero dejas de lado el proceso de aprendizaje de un lenguaje que, a largo plazo, impactará en tu capacidad de análisis y creatividad. Otra ventaja importante, es que de la misma manera en la que comunicas ideas al escribir un libro o un artículo, puedes escribir los análisis usando R. Esto permite dejar un registro completo de los procedimientos que se siguieron, permitiendo repetir los análisis meses o años después, además de poder compartirlos con otras personas.
Esta guía está dirigida a aquellas personas que quieren comenzar a utilizar R o a aquellas personas que, como nosotros, siempre olvidan algunas funciones y quisieran tener una ayuda a mano. En especial pensamos en estudiantes de licenciatura y en aquellos que comienzan su posgrado pero también estamos seguros que hay varios investigadores consagrados que quisieran comenzar a usar este lenguaje.
La guía está estructurada siguiendo las etapas generales del análisis de datos tales como: ingreso de datos, modificación de bases de datos, estadísticos básicos, gráficos y modelos lineales. No pretendemos cubrir exhaustivamente diferentes análisis estadísticos sino brindar una guía básica para el proceso de análisis usando R.
Finalmente, R es un software libre que puede ser copiado, distribuido y modificado a voluntad, lo que permite generar nuevas rutinas y paquetes de análisis. Esto te convierte en participante del desarrollo científico y no en un simple cliente que condiciona su creatividad a la paga de una licencia de un software cerrado, el cual evoluciona independientemente de las necesidades del usuario. La utilización de programas como R es una oportunidad que instituciones de caracter público y en especial de países en desarrollo, no deben dejar pasar.
Esperamos que esta versión wiki de la Guía de Campo de R sea de utilidad para cualquiera que quiera comenzar a trabajar con este lenguaje y de manera especial esperamos contribuir a la formación de los estudiantes e investigadores del Instituto de Ecología de la Universidad Nacional Autónoma de México y del Instituto Multidisciplinario de Biología Vegetal de la Universidad Nacional de Córdoba - CONICET, las instituciones donde trabajamos. Por ser esta una versión wiki esperamos que sufra modificaciones con el tiempo y estamos abiertos a sugerencias, correcciones y adiciones de nuevos capítulos por parte de cualquier interesado en colaborar con esta tarea. Los contenidos de esta guía son de libre difusión citando la fuente.
- Los autores
- Diego Carmona
- Santiago Benitez-Vieyra
INTRODUCCIÓN
Antes de empezar
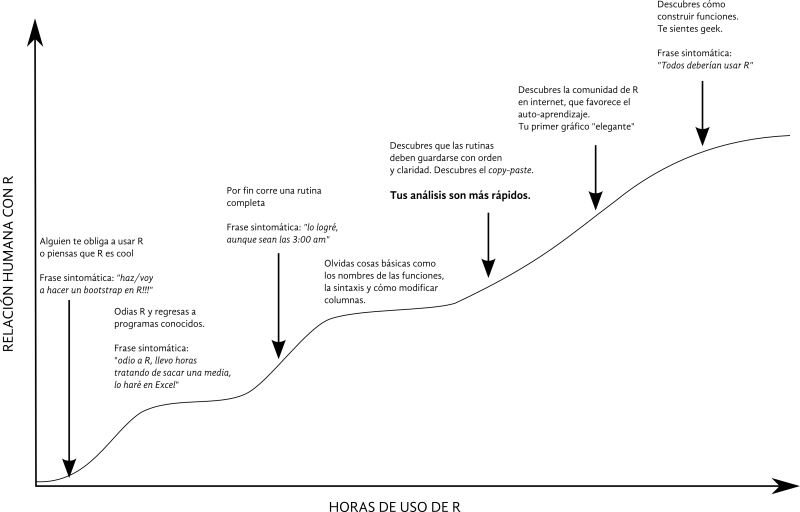
Creemos importante comenzar la guía resaltando la importancia de que lo que se va a aprender es un lenjuaje y como tal es un proceso el cual esta asociado a una curva de aprendizaje (Fig. 1). Tener esto en mente, pensamos, debería de disminuir la ansiedad al tratar de comunicarse con R. También, en esta sección te compartimos cuatro simbolos básicos para comenzar a entender la sintaxis de R.
Figura 1. Esta curva de aprendizaje muestra como aprender a usar R lleva tiempo y claramente impacta en nuestra relación cognitivo-emocional con el programa. Es muy probable que los saltos ocurran en diferente orden, lo relevante es notar el proceso.
| # | |
|---|---|
| #esto es una nota | El espacio a la derecha de este símbolo no es leído, por lo que sirve para poner anotaciones. |
| <- | |
a<-4+6 |
El símbolo asignar. Guarda el resultado de una operación dentro de un objeto llamado a en el ejemplo (el nombre es elegido arbitrariamente por el usuario). Si utilizo nuevamente ese nombre en una asignación (por ej a<-c(1:20)) el primer objeto se pierde. El símbolo = puede sustituir a <- (hay rutinas donde lo hacen así) pero esto podría resultar confuso en ciertos casos, por lo que no es recomendable. |
| ? | |
?mean ?lm ??"linear models" |
Si tienes duda sobre una función, puedes obtener información (poco amable, pero básica) usando el signo de interrogación. Si desconoces el nombre de la función que realizaría determinada acción, puedes hacer que R busque en todos los paquetes instalados usando ?? |
| c | |
vector<-c(1,2,3,4) vector<-c(1:4) vector<-c(“a”,”b”,”c”,”d”) vector<-c(“1”,”a”,”2”,”b”) |
C concatena (pega) elementos, ya sea numericos, categoricos. Es una de las funciones más básicas. |
Las ayudas para R
El símbolo ? sirve para acceder a las ayudas de cada una de las funciones de R, lo que requiere conocer el nombre de la función por anticipado. Cuando esto no sucede, la solución es sencillamente buscar el nombre del análisis que queremos hacer más “R” en internet (por ejemplo, “generalized linear models R”). En esta sección revisaremos las ayudas “oficiales” de R, las cuales pueden ser poco amigables. Sin embrago es crucial que utilices internet y que guardes las páginas que te resultaron útiles en una carpeta de marcadores o bookmarks en tu navegador. Algunas páginas de internet pueden ser de ayuda general, pero además existen muchas listas de correo donde los usuarios postean preguntas y respuestas. Casi siempre alguien ha tenido previamente el mismo problema que tu tienes, estas listas son el lugar ideal para buscar posibles soluciones.
Secciones de la ayuda de una función cualquiera.
- function {package}
- Esta parte identifica la función. El paquete debe estar cargado (ver sección 1.3) para que la ayuda funcione.
- Description.
- Breve descripción de lo que realiza la función. A veces es demasiado breve.
- Usage.
- Parte central de la ayuda. Toda función actúa sobre una serie de argumentos, los cuales se hallan encerrados entre paréntesis y separados por comas. En esta sección se enumeran todos los argumentos posibles (a veces es posible agregar argumentos adicionales no descriptos, en este caso se indica con puntos suspensivos). Estos argumentos se dividen en dos grupos, aquellos fijados en un valor por defecto (por ejemplo na.rm = FALSE, notar el símbolo igual) y aquellos que requieren que se especifique su valor (por ejemplo data). Estos últimos pueden ser optativos u obligatorios.
- Arguments.
- Descripción más detallada de los argumentos. Es muy importante que prestes atención al tipo de objeto (como marcos de datos, vectores, fórmulas o valores lógicos) que requiere cada argumento.
- Value.
- Especifica la salida de la función. En general cuando se realiza un análisis la función crea un objeto que guarda diferentes aspectos del resultado. Por ejemplo, si lo que realizamos es una regresión lineal y llamamos fit a nuestro análisis, los parámetros de la regresión estarán en fit$coeffcients, los residuales en fit$residuals, etc. Muchas veces no queremos conocer todos esos resultados, sino solamente un resumen. Esta actividad la realiza casi siempre la función summary.
- Las funciones gráficas solamente crean o modifican gráficos, en general no tienen salidas que mostrar.
- Author.
- El autor o autores de la función.
- References.
- Posibles libros o trabajos donde hay ayuda sobre el análisis que realiza la función.
- See Also.
- Lista de funciones relacionadas.
- Examples.
- Otra sección importante. Si copias y pegas el ejemplo en la consola de R, verás como trabaja la función. Es importante notar que los autores de los ejemplos en general incluyen una simulación de datos (para evitar que la ayuda tenga un set de datos que haya que ingresar cada vez), y que esta simulación a veces es confusa para los principiantes. Debes concentrarte en la parte donde se ejecuta la función.
Páginas de ayudas generales en R.
- http://www.r-project.org/
- http://www.r-bloggers.com/
- http://www.statmethods.net/index.html/
- http://rwiki.sciviews.org/doku.php
- http://r-forge.r-project.org/
- http://finzi.psych.upenn.edu/nmz.html
- http://www.rseek.org/
Sobre los paquetes y su instalación
Los objetos pueden ser creados, modificados o analizados con funciones básicas o complejas. Muchas de estas funciones ya están por defecto cargadas al abrir el programa. Otras miles se encuentran en paquetes específicos que son necesarios bajar de internet, instalarlos y cargarlos. Los paquetes se encuentran en repositorios (servidores o “espejos” que mantienen copias de todos los paquetes) al rededor del mundo y conviene utilizar aquellos que estén más cercanos a tu localidad.
| install.packages() | |
|---|---|
| install.packages ("car") | Te preguntará que “espejo” vas a utilizar para bajar el paquete. Una vez elegido comienza a descargar e instalar el paquete.
En windows o Rstudio se puede realizar desde el menu paquetes (o packages) |
| library() | |
| library(car) | Para usar el paquete es necesario previamente cargarlo (activarlo).
En windows Mac o Rstudio se puede realizar desde sus menus. |
Sobre los objetos
Cuando abres R por primera vez, la primera impresión es que no es amigable. Y en realidad no lo es. Los conocidos menús de Archivo, Editar, Ayuda, etc. están reducidos a su mínima expresión (o no existen en absoluto, si usas R en Ubuntu). Además, no sirven para nada "estadístico". No hay ventanitas para calcular la media, ajustar un modelo o pedir un análisis de componentes principales. Ni siquiera para hacer un gráfico. Para peor, probablemente el primer mensaje que R te dio es "Error: objeto X no encontrado". Todos estos problemas provienen de un rasgo fundamental: R es un lenguaje. Por lo tanto aprender R es como aprender inglés (o chino mandarín) y es muy diferente a aprender "recetas de cocina estadística".
Si bien R es un leguaje, no es muy eficiente para la poesía o para expresar emociones, pero es sumamente eficaz para contarle a otra persona los pasos de un análisis estadístico, realizar un gráfico o programar un nuevo método. Lo más importante es que cada vez que quieran repetir ese análisis o ese gráfico, los pasos para realizarlos ya estarán guardados en una rutina. Igualmente, es muy sencillo adaptar esa rutina a nuevos datos o modificarla para agregarle o quitarle algunos pasos.
La base de todo el lenguaje es el símbolo <- que crea objetos (en ocasiones podrás encontrar que se substituye por “=”, sin embargo preferimos la flechita por su directa relación con su significado, y para evitar posibles confusiones con otros usos de "="). Un objeto puede ser cualquier cosa: un set de datos, una columna de esos datos, una lista, una matriz, una fórmula, una función estadística o un resultado de un análisis. Es decir que, mientras usas el programa, un montón de esos objetos son creados y guardados en la memoria de la computadora. Por ejemplo, siguiendo uno de los pasos del gráfico de la siguiente sección (XXX), al importar datos se creará un objeto de tipo data.frame, sus columnas serán vectores (vectors), algunos de ellos serán factores (factor) y otros serán numéricos (numeric). Podemos construir una regresión con dos de estas columnas, aplicando la función lm. El resultado puede ser guardado en un objeto lm el cual es en realidad una lista (list) que en su interior, entre muchos otros, contiene distintos objetos como los coeficientes, los residuos, los predichos (todos ellos numeric), el número de observaciones (integer) y la fórmula usada (formula). De esta forma, puedes considerar que R es un espacio vacío abierto a ser llenado con objetos de diferentes naturalezas y propiedades y, allí, bajo ciertas reglas (que debes aprender), usar tu creatividad para hacerlos interactuar y crear tu propio mundo de análisis. De esta manera, podrás hacer rutinas tan sencillas como un tweeter o tan complejos y rebuscados como una novela. Sin embargo una buena programación es un arte de decir muchas cosas de manera sucinta como en un haiku. Sea cual sea la temática de tu programa es bueno tener la idea del flujo de trabajo, es decir, estructura.
| data.frame | Objeto que contiene nuestros datos. Habitualmente nuestros datos estarán almacenados en una tabla de Excel o Calc, y deberemos realizar un pequeño procedimiento para importarlos. Al realizar este procedimiento, un data.frame será creado. Ocasionalmente puede ser creado "a mano" dentro del mismo R.
Un data.frame consiste en una serie de variables (vectors) de igual longitud y que pueden ser del mismo tipo o no. |
| vector | Colección de datos del mismo tipo (números, letras, etc.) que puede ser creado dentro de R o importado como una columna de un data.frame. Existen muchos tipos de vectores, entre ellos:
numeric. Consiste de números reales (es decir que pueden tener decimales). integer. Consiste de números enteros. character. Contiene letras, nombres, etc. Notar que cada elemento se encuentra entre comillas (ej. "A1"). factor. Una forma más elegante de los character, sirve para representar variables categóricas, portando información extra sobre los niveles del factor (levels) y sobre si estos niveles siguen un orden o no. |
| matrix | Matriz formada por la unión de vectores de un mismo tipo y largo, por un solo vector que es partido en columnas y filas o (más habitualmente) producto de ciertas funciones, por ejemplo cor, que construye matrices de correlación. |
| list | Objeto que compuesto de objetos de distinto tipo y largo. |
Flujo del Trabajo en R
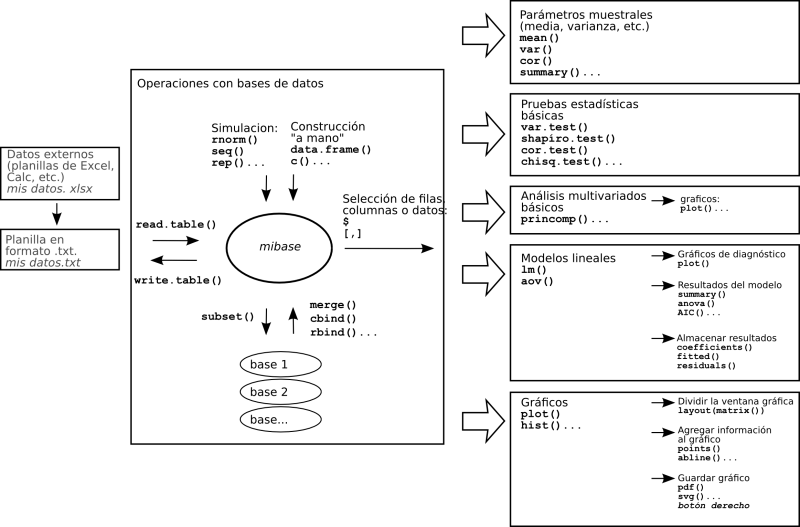
Los restantes capítulos de esta guía siguen el flujo de trabajo habitual en R. Comenzaremos por las distintas formas de ingresar conjuntos de datos, manipularlos, dividirlos en subgrupos o unir varios conjuntos de datos en uno solo. Una vez importados los datos podemos realizar diferentes operaciones sobre ellos, desde las más sencillas como extraer parámetros como la media o realizar una prueba t hasta realizar modelos lineales. La capacidad gráfica de R es particularmente poderosa, y una sección especial será dedicada a la realización de gráficos.
BASES DE DATOS
La base de datos a usar.
Construimos una base lo suficientemente general para ejemplificar las situaciones más comunes. La base tienen dos factores, población (pob) con tres poblaciones (i.e. tres niveles a, b y c) y tratamiento (trat) con dos niveles (1 y 2). Como podrás notar en una columna usamos letras y en otra números para denotar cada nivel del factor. R al identificar las letras sabrá que la variable es un factor, lo que no sucede si usas números. Por otro lado, la base tiene dos variables numéricas: tamaño (tam) que tiene una distribución normal y número de frutos (frutos) que tiene una distribución poisson (variables provenientes de conteos que no admiten números fraccionarios: por ejemplo 20 frutos, 6 frutos, etc.). Finalmente incluimos la columna obs que podría servir para identificar el individuo donde se hicieron las observaciones. Para cargar esta base de datos ve la sección siguiente. Puedes guardar la tabla copiando de la wiki a un block de notas o planilla de cálculo.
| obs | pob | trat | tam | frutos |
|---|---|---|---|---|
| 1 | a | 1 | 3.2246 | 10 |
| 2 | a | 1 | 2.3868 | 6 |
| 3 | a | 1 | NA | 5 |
| 4 | a | 1 | 2.7746 | 4 |
| 5 | a | 2 | 2.1741 | 8 |
| 6 | a | 2 | 1.844 | 6 |
| 7 | a | 2 | 1.6193 | 5 |
| 8 | a | 2 | 1.7563 | 12 |
| 9 | b | 1 | 3.1419 | 7 |
| 10 | b | 1 | 4.8306 | 12 |
| 11 | b | 1 | 4.1514 | 7 |
| 12 | b | 1 | 3.5897 | 8 |
| 13 | b | 2 | 3.4678 | 2 |
| 14 | b | 2 | 1.1311 | 4 |
| 15 | b | 2 | 3.0528 | 3 |
| 16 | b | 2 | 3.0423 | 6 |
| 17 | c | 1 | 3.6083 | 7 |
| 18 | c | 1 | 5.8285 | 11 |
| 19 | c | 1 | 4.5025 | 8 |
| 20 | c | 1 | 6.969 | 3 |
| 21 | c | 2 | 6.3181 | 10 |
| 22 | c | 2 | NA | 10 |
| 23 | c | 2 | 7.8368 | 20 |
| 24 | c | 2 | 7.5189 | 14 |
Importando y exportando bases de datos
Importando
El primer problema y quizás el más frustrante al comenzar a usar R es no poder ingresar los datos para comenzar los análisis. Una práctica que podría hacer más sencillo todo seria crear una única carpeta en tu computadora, con una ruta sencilla (es decir cerca de la "raíz" como C:/datos) donde siempre guardes archivos de datos (analisis R). Ej. C:/analisisR/tesis/datos.txt.
Tips: cosas que podrían producir problemas.
|
Para cargar la base de datos y bautizarla con el nombre de mibase se debe utilizar la función read.table. Al hacerlo, creamos un objeto (llamado aquí mibase) del tipo data.frame
| read.table / read.csv | |
|---|---|
|
mibase <- read.table("C:/analisisR/tesis/datos.txt", header = TRUE, sep = ".") #Windows mibase <- read.table("/Users/Yo/Documents/tesis/datos.txt", header = TRUE, sep = ".") #Mac mibase <- read.table("/home/Yo/Documentos/tesis/datos.txt", header = TRUE, sep = ".") #Ubuntu mibase <- read.csv("C:/analisisR/tesis/datos.csv", header = TRUE, sep = ".") #Windows |
Importar los datos, conociendo la ruta. Interpretación básica:
|
| file.choose() | |
|
mibase <- read.table(file=file.choose(),header=TRUE, sep = ".") |
Si uno no tiene clara la ruta. Contra: no puedes automatizar el cargar la base. |
| "clipboard" | |
|
mibase <- read.table("clipboard", header = TRUE, sep = ".") |
Método "quick & dirty" de importar datos: 1) Selecciona los datos en tu planilla y cópialos (pero no los pegues, dejalos en la memoria o sea en el portapapeles o clipboard); 2) Escribe esta rutina en R (no la copies). La rutina leerá el objeto que tengas en el portapapeles. 3) Recuerda presionar Enter. Si tienes problemas y no se pega bien el formato, podrias pegarlo primero en una tabla de excel y luego copiarlo de vuelta y entonces usar esta función para introducir los datos a R. |
| setwd() | |
|
setwd("C:/analisisR/tesis/") base1 <- read.table("datos1.txt", header = T) base2 <- read.table("datos2.txt", header = T) base3 <- read.table("datos3.txt", header = T) |
setwd fija la ruta que R usará para leer los archivos. Ventaja: evita escribir esa ruta cada vez, en cada read.table(). Desventaja: puedes olvidarte cuál era la ruta que usabas. Es ese caso usar getwd() |
Tips: importación de datos.
|
Exportando
Ocasionalmente querremos exportar nuestros datos de R hacia otro formato.
| write.table / write.csv | |
|---|---|
|
write.table(mibase, "c:/analisisR/tesis/mibase2.txt", col.names = TRUE, row.names = FALSE) write.csv(mibase, "c:/analisisR/tesis/mibase2.csv", col.names = TRUE, row.names = FALSE) |
Aquí se le da a R el nombre del objeto (i.e. mibase) que será salvado en un directorio particular con el nombre de mibase2 y con formato determinado (txt, csv, etc.) |
TRABAJANDO CON BASES DE DATOS
Los data.frame son un tipo de objeto muy común que consta de datos (objetos atómicos que por lo tanto ya no se pueden descomponer en más subobjetos) ordenados en filas (row) y columnas (col), cada una de ellas con el mismo número de filas. Cada uno de estos objetos tiene sus propiedades exclusivas que se verán en las próximas secciones. Por el momento mostraremos las propiedades del objeto data.frame. Los data.frame pueden contener columnas de diferente tipo, por ejemplo numéricas y categóricas.
Extrayendo información de un data.frame.
En la sección anterior cargamos una base de datos al cual llamamos mibase que debido a que fue cargada usando read.table es un objeto del tipo data.frame. En la siguiente tabla están las principales funciones para visualizar un set de datos y obtener información sobre él.
| escribir el nombre del objeto | |
|---|---|
|
mibase |
Escribir el nombre del objeto hace que R nos lo muestre de manera completa. Si es una base muy larga esto puede no ser muy buena idea. |
| head | |
| head(mibase) | Muy útil. Nos muestra las primeras seis filas de la base incluyendo los encabezados. El equivalente en indexación sería: mibase[1:6,]. |
| tail | |
| tail(mibase) | Nos muestra las últimas seis filas de la base. El equivalente en indexación sería: mibase[19:24,] (en este caso). |
| names | |
| names(mibase) | Permite ver los encabezados de todas las columnas. |
| nrow | |
| nrow(mibase) | Indica el número de filas únicamente |
| ncol | |
| ncol(mibase) | Indica el número de columnas únicamente |
| dim | |
| dim(mibase) | Indica las dimensiones de la base (# filas, # columnas) |
| na.omit | |
| mibase <- na.omit(mibase) | Quita todas las filas que contengan NA en la base de datos. Puede no ser muy recomendable hacer esto ya que uno puede perder mucha información (todas las filas con al menos un NA). |
| fix | |
| fix(mibase) | Abre una ventana donde pueden modificarse los datos. Los cambios no son guardados en el archivo de datos original, sino en el objeto data.frame que R guarda en la memoria, por lo que se pierden al cerrar el programa. No es muy recomendable su uso. |