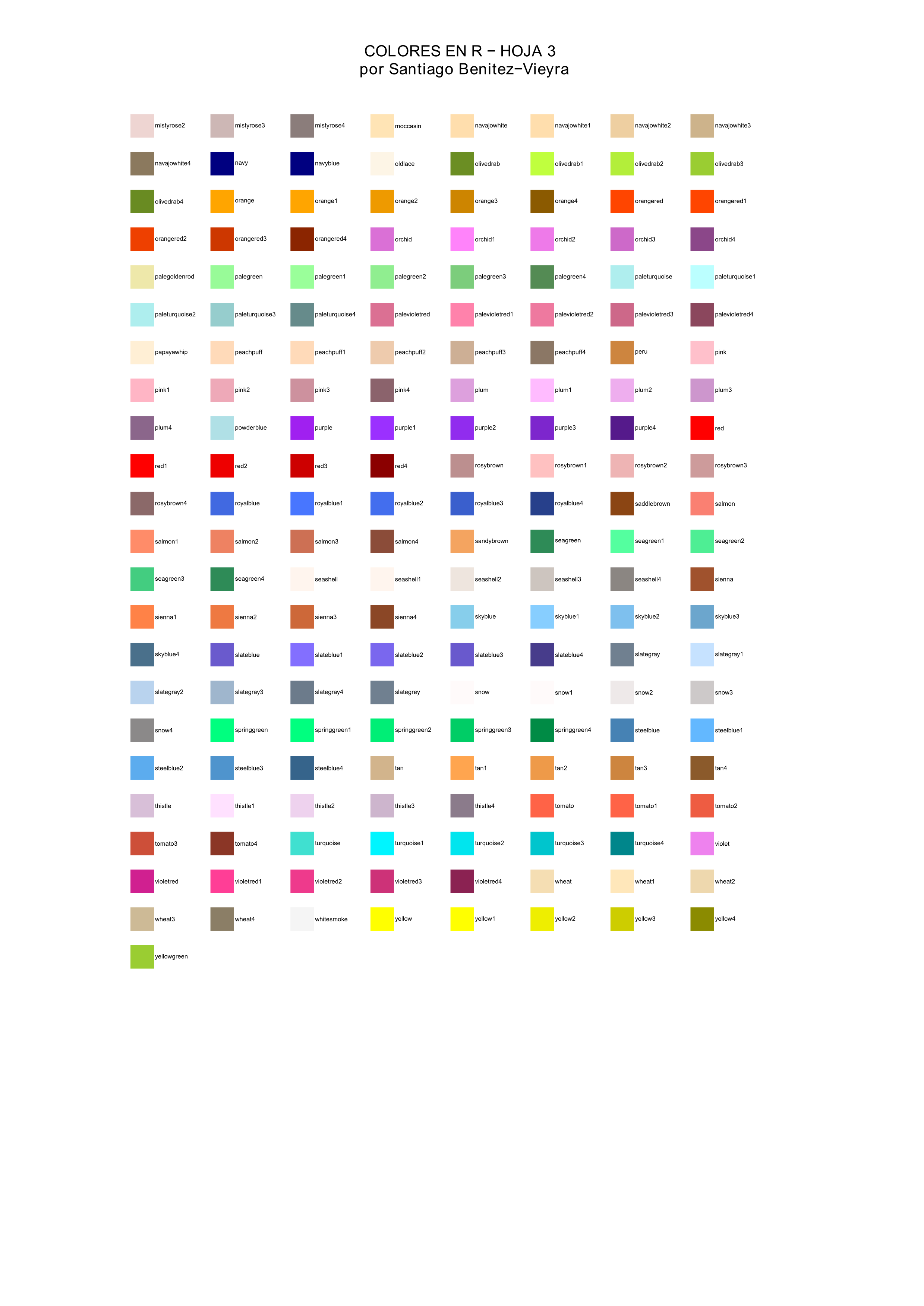Guía de campo de R
PRÓLOGO[editar]
Con esta guía buscamos aportar los fundamentos básicos para utilizar R de la manera más eficiente. R es un programa con grandes capacidades, sin embargo, en esta guía nos enfocamos en el manejo de bases de datos y análisis básicos. Como somos biólogos esta guía está sesgada al contexto de análisis de datos biológicos seguiendo la lógica de una guía de campo la cual, idealmente, debe ser concisa, liviana y bien ilustrada.
Para aquellos que comienzan a usar R, el manejo de bases de datos, la modificación de columnas y/o filas o la obtención de estadísticas básicas son uno de los principales retos. Muchas veces los principiantes se ven tentados a regresar a los viejos programas y dejan R para el momento de hacer análisis "sofisticados". Esta práctica es en realidad muy poco eficiente y cortoplacista ya que obtienes lo necesario en el momento pero dejas de lado el proceso de aprendizaje de un lenguaje que, a largo plazo, impactará en tu capacidad de análisis y creatividad. Otra ventaja importante es que, de la misma manera en que comunicas ideas al escribir un libro o un artículo, puedes escribir los análisis usando R. Esto permite dejar un registro completo de los procedimientos que se siguieron, permitiendo repetir los análisis meses o años después, además de poder compartirlos con otras personas.
Esta guía está dirigida a aquellas personas que quieren comenzar a utilizar R o a aquellas personas que, como nosotros, siempre olvidan algunas funciones y quisieran tener una ayuda a mano. En especial pensamos en estudiantes de licenciatura y en aquellos que comienzan su posgrado, pero también estamos seguros que hay varios investigadores consagrados que quisieran comenzar a usar este lenguaje.
La guía está estructurada siguiendo las etapas generales del análisis de datos tales como ingreso de datos, modificación de bases de datos, estadísticos básicos, gráficos y modelos lineales. No pretendemos cubrir exhaustivamente diferentes análisis estadísticos sino brindar una guía básica para el proceso de análisis usando R.
Finalmente, R es un software libre que puede ser copiado, distribuido y modificado a voluntad, lo que permite generar nuevas rutinas y paquetes de análisis. Esto te convierte en participante del desarrollo científico y no en un simple cliente que condiciona su creatividad a la paga de una licencia de un software cerrado, el cual evoluciona independientemente de las necesidades del usuario. La utilización de programas como R es una oportunidad que instituciones de caracter público y en especial de países en desarrollo, no deben dejar pasar.
Esperamos que esta versión wiki de la Guía de Campo de R sea de utilidad para cualquiera que quiera comenzar a trabajar con este lenguaje y de manera especial esperamos contribuir a la formación de los estudiantes e investigadores del Instituto de Ecología de la Universidad Nacional Autónoma de México y del Instituto Multidisciplinario de Biología Vegetal de la Universidad Nacional de Córdoba - CONICET, las instituciones donde trabajamos. Por ser esta una versión wiki esperamos que sufra modificaciones con el tiempo y estamos abiertos a sugerencias, correcciones y adiciones de nuevos capítulos por parte de cualquier interesado en colaborar con esta tarea. Los contenidos de esta guía son de libre difusión citando la fuente.
- Los autores
- Diego Carmona
- Santiago Benitez-Vieyra
INTRODUCCIÓN[editar]
Antes de empezar[editar]
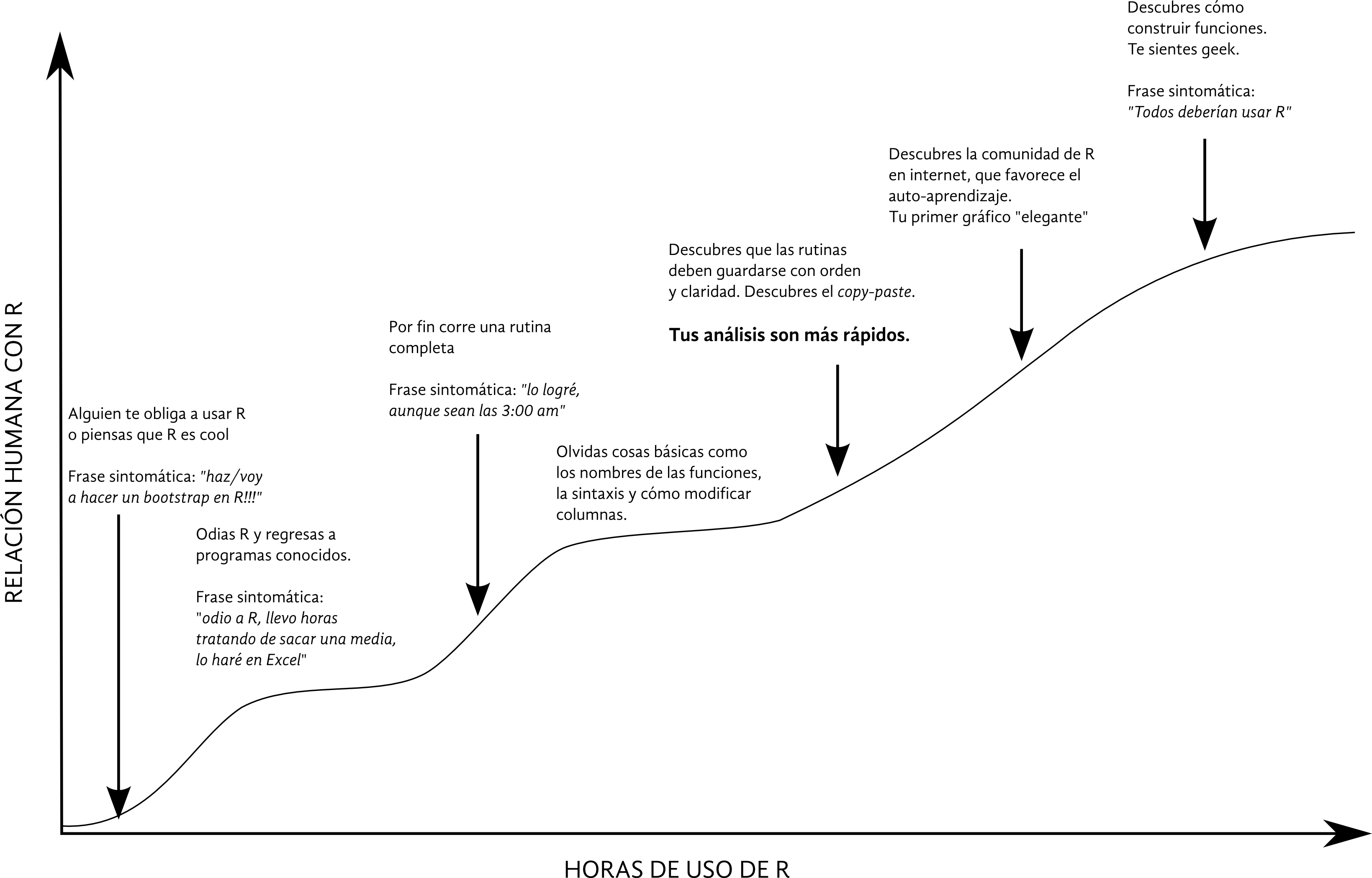
Creemos importante comenzar la guía resaltando que R es un lenguaje y que su aprendizaje, como todos, se halla asociado a una curva (Fig. 1). Tener esto en mente, pensamos, debería de disminuir la ansiedad al tratar de comunicarse con R. También, en esta sección te compartimos cuatro símbolos básicos para comenzar a entender la sintaxis de R.
Esta curva de aprendizaje muestra como aprender a usar R lleva tiempo y claramente impacta en nuestra relación cognitivo-emocional con el programa. Es muy probable que los saltos ocurran en diferente orden, lo relevante es notar el proceso.
| # | |
|---|---|
|
# esto es una nota |
El espacio a la derecha de este símbolo no es leído, por lo que sirve para poner anotaciones. |
| <- | |
|
a <- 4 + 6 |
El símbolo asignar. Guarda el resultado de una operación dentro de un objeto llamado a en el ejemplo (el nombre es elegido arbitrariamente por el usuario). Si utilizo nuevamente ese nombre en una asignación (por ej a<-c(1:20)) el primer objeto se pierde. El símbolo = puede sustituir a <- (hay rutinas donde lo hacen así) pero esto podría resultar confuso en ciertos casos, por lo que no es recomendable. |
| ? | |
|
?mean ?lm ??"linear models" |
Si tienes duda sobre una función, puedes obtener información (poco amable, pero básica) usando el signo de interrogación. Si desconoces el nombre de la función que realizaría determinada acción, puedes hacer que R busque en todos los paquetes instalados usando ?? |
| c | |
|
vector <- c(1, 2, 3, 4) vector <- c(1:4) vector <- c(“a”, ”b”, ”c”, ”d”) vector <- c(“1”, ”a”, ”2”, ”b”) |
c concatena (pega) elementos, ya sea numéricos, categóricos. Es una de las funciones más básicas. |
Las ayudas para R[editar]
El símbolo ? sirve para acceder a las ayudas de cada una de las funciones de R, lo que requiere conocer el nombre de la función por anticipado. Cuando esto no sucede, la solución es sencillamente buscar el nombre del análisis que queremos hacer más “R” en internet (por ejemplo, “generalized linear models R”). En esta sección revisaremos las ayudas “oficiales” de R, las cuales pueden ser poco amigables. Sin embargo es crucial que utilices internet y que guardes las páginas que te resultaron útiles como marcadores o bookmarks en tu navegador. Algunas páginas de internet pueden ser de ayuda general, pero además existen muchas listas de correo donde los usuarios postean preguntas y respuestas. Casi siempre alguien ha tenido previamente el mismo problema que tu tienes, estas listas son el lugar ideal para buscar posibles soluciones.
Secciones de la ayuda de una función cualquiera.
- function {package}
- Esta parte identifica la función. El paquete debe estar cargado (ver sección 1.3) para que la ayuda funcione.
- Description.
- Breve descripción de lo que realiza la función. A veces es demasiado breve.
- Usage.
- Parte central de la ayuda. Toda función actúa sobre una serie de argumentos, los cuales se hallan encerrados entre paréntesis y separados por comas. En esta sección se enumeran todos los argumentos posibles (a veces es posible agregar argumentos adicionales no descriptos, en este caso se indica con puntos suspensivos). Estos argumentos se dividen en dos grupos, aquellos fijados en un valor por defecto (por ejemplo na.rm = FALSE, notar el símbolo igual) y aquellos que requieren que se especifique su valor (por ejemplo data). Estos últimos pueden ser optativos u obligatorios.
- Arguments.
- Descripción más detallada de los argumentos. Es muy importante que prestes atención al tipo de objeto (como marcos de datos, vectores, fórmulas o valores lógicos) que requiere cada argumento.
- Value.
- Especifica la salida de la función. En general, cuando se realiza un análisis estadístico, la función crea un objeto que guarda diferentes aspectos del resultado. Por ejemplo, si lo que realizamos es una regresión lineal y llamamos fit a nuestro análisis, los parámetros de la regresión estarán en fit$coeffcients, los residuales en fit$residuals, etc. Muchas veces no queremos conocer todos esos resultados, sino solamente un resumen. Esta actividad la realiza casi siempre la función summary.
- Las funciones gráficas solamente crean o modifican gráficos, en general no tienen valores que mostrar.
- Author.
- El autor o autores de la función.
- References.
- Posibles libros o trabajos donde hay ayuda sobre el análisis que realiza la función.
- See Also.
- Lista de funciones relacionadas.
- Examples.
- Otra sección importante. Si copias y pegas el ejemplo en la consola de R, verás como trabaja la función. Es importante notar que los autores de los ejemplos en general incluyen una simulación de datos (para evitar que la ayuda tenga un set de datos que haya que ingresar cada vez), y que esta simulación a veces es confusa para los principiantes. Debes concentrarte en la parte donde se ejecuta la función.
Páginas de ayudas generales para R.
- http://www.r-project.org/
- http://www.r-bloggers.com/
- http://www.statmethods.net/index.html/
- http://rwiki.sciviews.org/doku.php
- http://r-forge.r-project.org/
- http://finzi.psych.upenn.edu/nmz.html
- http://www.rseek.org/
Sobre los paquetes y su instalación[editar]
Los objetos pueden ser creados, modificados o analizados con funciones básicas o complejas. Muchas de estas funciones ya están por defecto cargadas al abrir el programa. Otras miles se encuentran en paquetes específicos que son necesarios bajar de internet, instalarlos y cargarlos. Los paquetes se encuentran en repositorios (servidores o “espejos” que mantienen copias de todos los paquetes) alrededor del mundo y conviene utilizar aquellos que estén más cercanos a tu localidad.
| install.packages() | |
|---|---|
|
install.packages("car") |
Te preguntará que “espejo” vas a utilizar para bajar el paquete. Una vez elegido comienza a descargar e instalar el paquete. En windows o Rstudio se puede realizar desde el menu paquetes (o packages) |
| library() | |
|
library(car) |
Para usar el paquete es necesario previamente cargarlo (activarlo). En Windows, Mac o Rstudio se puede realizar desde sus menús. Sin embargo, cuando realices una rutina es recomendable incluir al principio los comandos de carga de todos los paquetes que vayas a utilizar. |
Sobre los objetos[editar]
Cuando abres R por primera vez, la primera impresión es que no es amigable. Y en realidad no lo es. Los conocidos menús de Archivo, Editar, Ayuda, etc. están reducidos a su mínima expresión (o no existen en absoluto, si usas R en Ubuntu). Además, no sirven para nada "estadístico". No hay ventanitas para calcular la media, ajustar un modelo o pedir un análisis de componentes principales. Ni siquiera para hacer un gráfico. Para peor, probablemente el primer mensaje que R te dio es "Error: objeto X no encontrado". Todos estos problemas provienen de un rasgo fundamental: R es un lenguaje. Por lo tanto aprender R es como aprender inglés (o chino mandarín) y es muy diferente a aprender "recetas de cocina estadística".
Si bien R es un lenguaje, no es muy eficiente para la poesía o para expresar emociones, pero es sumamente eficaz para contarle a otra persona los pasos de un análisis estadístico, realizar un gráfico o programar un nuevo método. Lo más importante es que cada vez que quieran repetir ese análisis o ese gráfico, los pasos para realizarlos ya estarán guardados en una rutina. Igualmente, es muy sencillo adaptar esa rutina a nuevos datos o modificarla para agregarle o quitarle algunos pasos.
La base de todo el lenguaje es el símbolo <- que crea objetos (en ocasiones podrás encontrar que se substituye por “=”, sin embargo preferimos la flechita por su directa relación con su significado, y para evitar posibles confusiones con otros usos de "="). Un objeto puede ser cualquier cosa: un set de datos, una columna de esos datos, una lista, una matriz, una fórmula, una función estadística o un resultado de un análisis. Es decir que, mientras usas el programa, un montón de esos objetos son creados y guardados en la memoria de la computadora. Por ejemplo, siguiendo uno de los pasos del gráfico de la siguiente sección, al importar datos se creará un objeto de tipo data.frame, sus columnas serán vectores (vectors), algunos de ellos serán factores (factor) y otros serán numéricos (numeric). Podemos construir una regresión con dos de estas columnas, aplicando la función lm. El resultado puede ser guardado en un objeto lm el cual es en realidad una lista (list) que en su interior, entre muchos otros, contiene distintos objetos como los coeficientes, los residuos, los predichos (todos ellos numeric), el número de observaciones (integer) y la fórmula usada (formula). De esta forma, puedes considerar que R es un espacio vacío abierto a ser llenado con objetos de diferentes naturalezas y propiedades y, allí, bajo ciertas reglas (que debes aprender), usar tu creatividad para hacerlos interactuar y crear tu propio mundo de análisis. De esta manera, podrás hacer rutinas tan sencillas como un tweet o tan complejos y rebuscados como una novela. Sin embargo una buena programación es un arte de decir muchas cosas de manera sucinta como en un haiku. Sea cual sea la temática de tu programa es bueno tener la idea del flujo de trabajo, es decir, estructura.
| data.frame | Objeto que contiene nuestros datos. Habitualmente nuestros datos estarán almacenados en una tabla de Excel o Calc, y deberemos realizar un pequeño procedimiento para importarlos. Al realizar este procedimiento, un data.frame será creado. Ocasionalmente puede ser creado "a mano" dentro del mismo R.
Un data.frame consiste en una serie de variables (vectors) de igual longitud y que pueden ser del mismo tipo o no. |
|
vector |
Colección de datos del mismo tipo (números, letras, etc.) que puede ser creado dentro de R o importado como una columna de un data.frame. Existen muchos tipos de vectores, entre ellos:
numeric. Consiste de números reales (es decir que pueden tener decimales). integer. Consiste de números enteros. character. Contiene letras, nombres, etc. Notar que cada elemento se encuentra entre comillas (ej. "A1"). factor. Una forma más elegante de los character, sirve para representar variables categóricas, portando información extra sobre los niveles del factor (levels) y sobre si estos niveles siguen un orden o no. |
|
matrix |
Matriz formada por la unión de vectores de un mismo tipo y largo, por un solo vector que es partido en columnas y filas o (más habitualmente) producto de ciertas funciones, por ejemplo cor, que construye matrices de correlación. |
|
list |
Objeto que compuesto de objetos de distinto tipo y largo. |
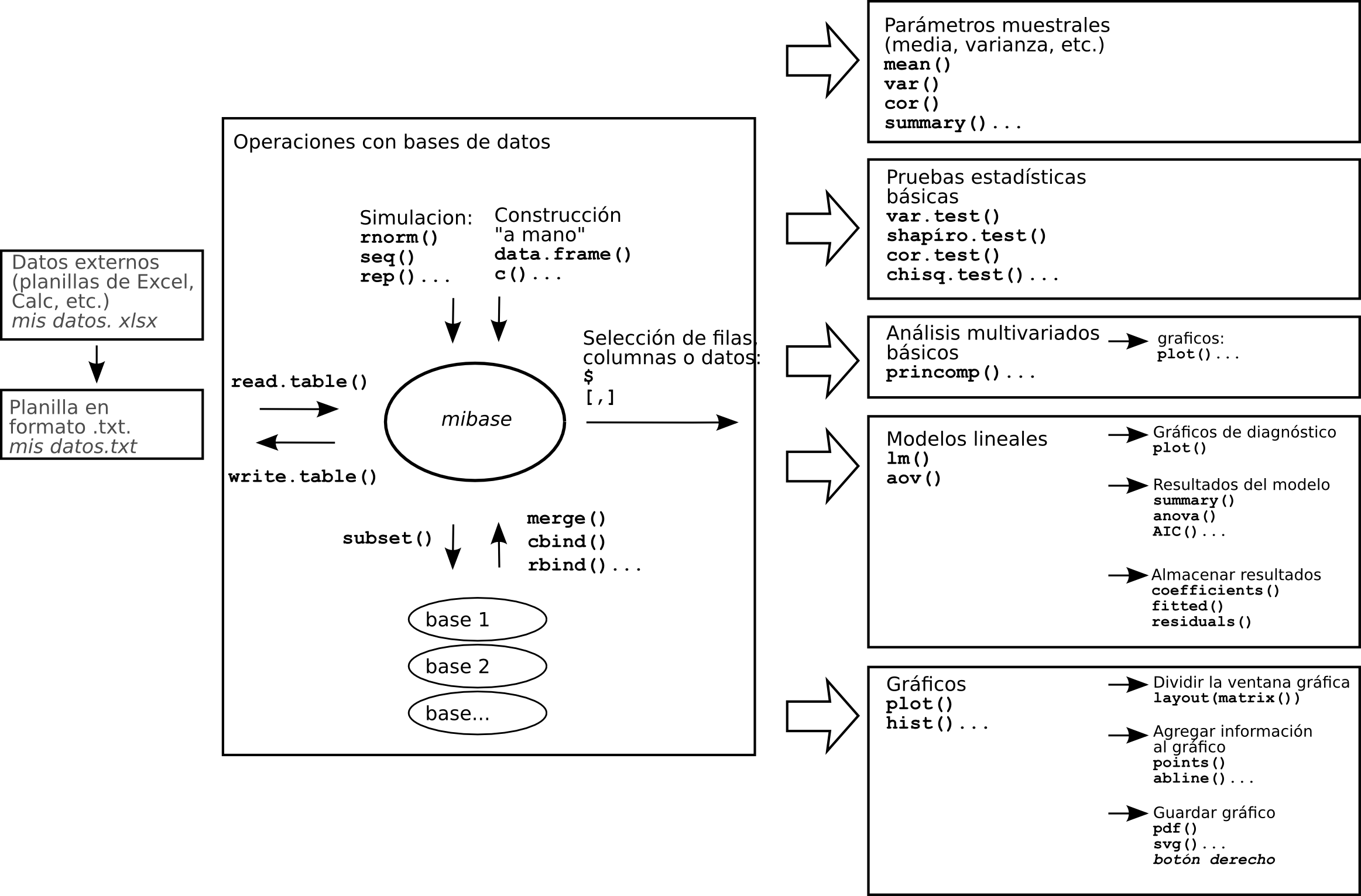
Flujo del Trabajo en R[editar]
Los restantes capítulos de esta guía siguen el flujo de trabajo habitual en R. Comenzaremos por las distintas formas de ingresar conjuntos de datos, manipularlos, dividirlos en subgrupos o unir varios conjuntos de datos en uno solo. Una vez importados los datos podemos realizar diferentes operaciones sobre ellos, desde las más sencillas como extraer parámetros como la media o realizar una prueba t hasta realizar modelos lineales. La capacidad gráfica de R es particularmente poderosa, y una sección especial será dedicada a la realización de gráficos.
Sobre las rutinas[editar]
Muchas veces desde el inicio de la guía hemos nombrado las "rutinas", pero ¿qué son esas rutinas? Una rutina es cualquier anotación que contenga los comandos para realizar un análisis, un gráfico o lo que sea en R. Sólo contiene los comandos (instrucciones) pero no la salida (resultados). Lo más útil es guardarla en un archivo de manera de poder cargarla y ejecutarla directamente en R (usando el formato .R) o al menos que se pueda ejecutar haciendo "copy-paste".
Es recomendable utilizar un texto "plano" y una tipografía de igual ancho para todas las letras (es decir, donde una "i" ocupe el mismo espacio que una "A", como Courier New). Jamás utilices Word para construirla, o sufrirás al corrector ortográfico y gramatical metiendo sus garras donde no debe. Utiliza el editor del mismo R o Block de Notas. Muchos programas además reconocen el "lenguaje R" y asignan diferentes colores a los signos y argumentos de manera de facilitar la escritura. Entre ellos sobresalen gedit, Tinn R y R Studio. Todos ellos son libres, y el último ofrece además muchas utilidades para interactuar con R.
BASES DE DATOS[editar]
La base de datos a usar[editar]
Construimos una base lo suficientemente general para ejemplificar las situaciones más comunes. La base tienen dos factores, población (pob) con tres poblaciones (i.e. tres niveles a, b y c) y tratamiento (trat) con dos niveles (1 y 2). Como podrás notar en una columna usamos letras y en otra números para denotar cada nivel del factor. R al identificar las letras sabrá que la variable es un factor, lo que no sucede si usas números. Por otro lado, la base tiene dos variables numéricas: tamaño (tam) que tiene una distribución normal y número de frutos (frutos) que tiene una distribución poisson (variables provenientes de conteos que no admiten números fraccionarios: por ejemplo 20 frutos, 6 frutos, etc.). Finalmente incluimos la columna obs que podría servir para identificar el individuo donde se hicieron las observaciones. Para cargar esta base de datos ve la sección siguiente. Puedes guardarla en una tabla copiando de la wiki a un block de notas o planilla de cálculo.
mibase
| obs | pob | trat | tam | frutos |
|---|---|---|---|---|
| 1 | a | 1 | 3.2246 | 10 |
| 2 | a | 1 | 2.3868 | 6 |
| 3 | a | 1 | NA | 5 |
| 4 | a | 1 | 2.7746 | 4 |
| 5 | a | 2 | 2.1741 | 8 |
| 6 | a | 2 | 1.844 | 6 |
| 7 | a | 2 | 1.6193 | 5 |
| 8 | a | 2 | 1.7563 | 12 |
| 9 | b | 1 | 3.1419 | 7 |
| 10 | b | 1 | 4.8306 | 12 |
| 11 | b | 1 | 4.1514 | 7 |
| 12 | b | 1 | 3.5897 | 8 |
| 13 | b | 2 | 3.4678 | 2 |
| 14 | b | 2 | 1.1311 | 4 |
| 15 | b | 2 | 3.0528 | 3 |
| 16 | b | 2 | 3.0423 | 6 |
| 17 | c | 1 | 3.6083 | 7 |
| 18 | c | 1 | 5.8285 | 11 |
| 19 | c | 1 | 4.5025 | 8 |
| 20 | c | 1 | 6.969 | 3 |
| 21 | c | 2 | 6.3181 | 10 |
| 22 | c | 2 | NA | 10 |
| 23 | c | 2 | 7.8368 | 20 |
| 24 | c | 2 | 7.5189 | 14 |
Importando y exportando bases de datos[editar]
Importando[editar]
El primer problema y quizás el más frustrante al comenzar a usar R es no poder ingresar los datos para comenzar los análisis. Una práctica que podría hacer más sencillo todo el proceso sería crear una única carpeta en tu computadora, con una ruta sencilla (es decir cerca de la "raíz" como C:/datos) donde siempre guardes archivos de datos (analisis R). Ej. C:/analisisR/tesis/datos.txt.
Tips: cosas que podrían producir problemas.
|
Para cargar la base de datos y bautizarla con el nombre de mibase se debe utilizar la función read.table. Al hacerlo, creamos un objeto (llamado aquí mibase) del tipo data.frame
| |read.table / read.csv | |
|---|---|
|
mibase <- read.table("C:/analisisR/tesis/datos.txt", header = TRUE, sep = ".") #Windows mibase <- read.table("/Users/Yo/Documents/tesis/datos.txt", header = TRUE, sep = ".") #Mac mibase <- read.table("/home/Yo/Documentos/tesis/datos.txt", header = TRUE, sep = ".") #Ubuntu mibase <- read.csv("C:/analisisR/tesis/datos.csv", header = TRUE, sep = ".") #Windows |
Importar los datos, conociendo la ruta. Interpretación básica:
|
| file.choose() | |
|
mibase <- read.table(file=file.choose(),header=TRUE, sep = ".") |
Si uno no tiene clara la ruta. Contra: no puedes automatizar el cargar la base. |
| "clipboard" | |
|
mibase <- read.table("clipboard", header = TRUE, sep = ".") |
Método "quick & dirty" de importar datos: 1) Selecciona los datos en tu planilla y cópialos (pero no los pegues, dejalos en la memoria o sea en el portapapeles o clipboard); 2) Escribe esta rutina en R (no la copies). La rutina leerá el objeto que tengas en el portapapeles. 3) Recuerda presionar Enter. Si tienes problemas y no se pega bien el formato, podrias pegarlo primero en una tabla de Excel y luego copiarlo de vuelta y entonces usar esta función para introducir los datos a R. |
| setwd() | |
|
setwd("C:/analisisR/tesis/") base1 <- read.table("datos1.txt", header = T) base2 <- read.table("datos2.txt", header = T) base3 <- read.table("datos3.txt", header = T) |
setwd fija la ruta que R usará para leer los archivos. Ventaja: evita escribir esa ruta cada vez, en cada read.table(). Desventaja: puedes olvidarte cuál era la ruta que usabas. Es ese caso usar getwd() |
Tips: importación de datos.
|
Exportando[editar]
Ocasionalmente querremos exportar nuestros datos de R hacia otro formato.
| write.table / write.csv | |
|---|---|
|
write.table(mibase, "C:/analisisR/tesis/mibase2.txt", col.names = TRUE, row.names = FALSE) write.csv(mibase, "C:/analisisR/tesis/mibase2.csv", col.names = TRUE, row.names = FALSE) |
Aquí se le da a R el nombre del objeto (i.e. mibase) que será salvado en un directorio particular con el nombre de mibase2 y con formato determinado (txt, csv, etc.) |
TRABAJANDO CON BASES DE DATOS[editar]
Los data.frame son un tipo de objeto muy común que consta de datos (objetos atómicos que por lo tanto ya no se pueden descomponer en más subobjetos) ordenados en filas (row) y columnas (col), cada una de ellas con el mismo número de filas. Cada uno de estos objetos tiene sus propiedades exclusivas que se verán en las próximas secciones. Por el momento mostraremos las propiedades del objeto data.frame. Los data.frame pueden contener columnas de diferente tipo, por ejemplo numéricas y categóricas.
Extrayendo información de un data.frame[editar]
En la sección anterior cargamos una base de datos al cual llamamos mibase que debido a que fue cargada usando read.table es un objeto del tipo data.frame. En la siguiente tabla están las principales funciones para visualizar un set de datos y obtener información sobre él.
| escribir el nombre del objeto | |
|---|---|
|
mibase |
Escribir el nombre del objeto hace que R nos lo muestre de manera completa. Si es una base muy larga esto puede no ser muy buena idea. |
| head | |
|
head(mibase) |
Muy útil. Nos muestra las primeras seis filas de la base incluyendo los encabezados. El equivalente en indexación sería: mibase[1:6,]. |
| tail | |
|
tail(mibase) |
Nos muestra las últimas seis filas de la base. El equivalente en indexación sería: mibase[19:24, ] (en este caso). |
| names | |
|
names(mibase) |
Permite ver los encabezados de todas las columnas. |
| nrow | |
|
nrow(mibase) |
Indica el número de filas únicamente |
| ncol | |
|
ncol(mibase) |
Indica el número de columnas únicamente |
| dim | |
|
dim(mibase) |
Indica las dimensiones de la base (# filas, # columnas) |
| na.omit | |
|
mibase <- na.omit(mibase) |
Quita todas las filas que contengan NA en la base de datos. Puede no ser muy recomendable hacer esto ya que uno puede perder mucha información (todas las filas con al menos un NA). |
| fix | |
|
fix(mibase) |
Abre una ventana donde pueden modificarse los datos. Los cambios no son guardados en el archivo de datos original, sino en el objeto data.frame que R guarda en la memoria, por lo que se pierden al cerrar el programa. No es muy recomendable su uso. |
Accediendo a la información contenida en un data.frame[editar]
Para acceder tanto a las filas como a las columnas hay varias formas de hacerlo y cada una tiene su propia ventaja. La forma más clara de entender que las columnas y las filas están anidadas en el data.frame es por medio de la indexación. Éstos son métodos aplicables a varios tipos de objetos y permiten una manipulación de los objetos muy fina.
| [ , ] | |
|---|---|
| mibase[row, col] | Ventaja: sintaxis general aplicable a cualquier tipo de objeto no solo a data.frame. |
| mibase[1:18, ] | Muestra las filas del 1 al 18 y todas las columnas del objeto mibase. |
|
mibase[, 1:4] |
Muestra las columnas del 1 al 4 y todas las filas del objeto mibase. |
|
mibase[1:18, 1:4] |
Muestra todas las filas y todas las columnas del objeto mibase. |
|
mibase [c(1:6, 8:18), 1:3] |
Muestra las filas 1 a 6, omite la fila 7 y luego muestra de la 8 a la 18. Mientras tanto, solo muestra las primeras 3 columnas. c indica concatenación (unir) la secuencia 1 a 6 con la secuencia 8 a 18. |
| mibase[-c(1:18), -2] | Muestra todas las filas, excepto aquellas del 1 al 18, y todas las columnas, excepto la segunda |
| $ | |
|
mibase$pob |
Muestra la columna llamada pob que se encuentra en el objeto mibase. |
| combinando $ y [ ] | |
|
mibase$tam [1:18] |
Muestra los elementos 1 al 18 de la columna tam. |
|
mibase$tam [-7] |
Excluye el séptimo elemento de la columna tam. |
|
mibase$tam [mibase$tam < 2] |
Muestra los elementos de la columna tam menores a 2. Claro también se puede usar ">" e "==" (significa =) y “!=” (que significa distinto a ...) |
Trabajando con columnas[editar]
La información de una columna[editar]
Una base de datos suele tener varias columnas y cada una de ellas tener sus propias particularidades. Muchas veces necesitamos conocer qué tipo de datos almacena una columna, crear nuevas o transformarlas.
| combinando $ y [ , ] | |
|---|---|
|
mibase$tam mibase[, "pob"] |
Mostrar una columna. Notar que si esta columna es un factor, al final indicará sus niveles (levels: a, b, c; en el caso de pob) |
| class | |
|
class (mibase$pob) class (mibase$frutos) |
Hay dos tipos columnas principales dependiendo de qué tipo de objetos contiene: numeric o factor. Los factores a su vez pueden estar ordenados o no ordenados y cada uno de sus elementos consiste en un character. OJO: El no tener bien definido el tipo de datos con el que se quiere trabajar es uno de los principales problemas para correr ciertos análisis. |
| levels | |
|
levels (mibase$pob) |
Te confirma el número de niveles que tiene esta factor y te indica el orden. |
| is... | |
|
is.numeric(mibase$tam) is.factor(mibase$pob) is.factor(mibase$frutos) |
Se le puede preguntar explícitamente si esa columna es numérica o factor y si así es R responde con TRUE.
Si tiene letras la columna por defecto será factor. En una columna numérica las únicas letras aceptadas son NA. Si usaste un símbolo decimal equivocado en read.table, R leerá todos tus números como letras. |
| length | |
|
length(mibase$tam) |
Determinando la longitud de la columna. Por ser una columna obviamente no tiene ancho. |
| which | |
|
which (is.na(mibase$tam)) length (which(is.na(mibase$tam))) |
Te indica que elementos de una columna poseen, en este caso, un NA.Combinado con length indica cuantos elementos cumplen esa condición. |
Creando nuevas columnas[editar]
En realidad, una columna es un vector con cierto número de objetos con un orden determinado. Por ejemplo, la columna obs es un vector que va de 1 a 24 mientras que la columna pob repite ocho veces “a”, ocho veces “b” y ocho veces “c”. Aprender a hacer vectores nos da mucho poder para crear nuevas formas de categorizar nuestra base de datos. Esto podrá tener grandes beneficios a la hora de manipular la base e incluso de analizarla.
| <- | |
|---|---|
|
mibase$log.frutos <- log(mibase$frutos) # logaritmo natural mibase$log.frutos <- log(mibase$frutos, 10) # logaritmo base 10 mibase$z.tam <- scale(mibase$tam) # estandarizar mibase$tam <- log(mibase$tam) # "pisar variables" |
Agregar una nueva columna es tan fácil como inventar un nombre y agregarle contenido con una función. Nota que si uso el mismo nombre, estoy reemplazando valores ("pisando"), y los valores originales se pierden. |
| c | |
|
mibase$obs <- c(1:24) |
Indica concatenar. Los dos puntos evitan escribir todos los números intermedios. De esta forma construimos la columna obs. Si en vez de números consecutivos quieres usar otros valores, hay que escribir cada uno de ellos, separados por coma. |
| rep | |
|
mibase$rep <- rep(c(rep("a", 12), rep("b", 12))) |
Creando columna rep La primera rutina se lee así: repite "a" 12 veces y luego "b" 12 veces. Luego concatena esta secuencia. Importante, el principal error en este tipo de sintaxis son los paréntesis, se cuidadoso. |
| seq | |
|
mibase$seq <- seq (0, 1, length.out = 24) |
Hace una secuencia del 0 al 1 con 24 pasos. |
| paste | |
|
mibase$clave <- paste(mibase$pob, mibase$trat, sep = "") mibase$clave <- paste(mibase$pob, mibase$trat, sep = ".") |
Uniendo factores para crear una clave más complicada. Une pob y trat sin separarlos con nada (sep="") por lo que quedara a1, a2,…,c8. Si se desea separar con un carácter como el punto se hace sep="." Esto da como resultado a.1,a.2,…,c.8. |
| strsplit | |
|
strsplit(mibase$clave, split = "") strsplit(mibase$clave, split = ";") strsplit(mibase$clave, split = "\\.") H <- strsplit(mibase$clave, split = "") pob <-numeric(24) for (i in 1:24) pob[i]<-H[[i]][1] pob |
Invierte la función anterior. Nos permite separar los caracteres y los muestra en forma de lista, un tipo de objeto un tanto incomodo. Si el separador fue punto requiere una notación particular. Para volver a un objeto cómodo se requieren pasos de programación que involucran usar for. |
Modificando una columna[editar]
Las situaciones más comunes en las cuales hay que modificar el contenido de una columna son: 1) cuando codificamos una variable categórica con números y debemos avisarle a R que los lea como letras (si, por ejemplo, planeamos hacer un ANOVA, ver sección XXX); 2) cuando queremos crear una nueva columna con datos transformados (por ejemplo tomando logaritmos o estandarizándolos).
| colnames | |
|---|---|
| colnames(mibase)[3] <- "edad" | Para cambiar de nombre la columna 3 de trat a edad |
| colnames(mibase) <- c("observ", "población", "tratamiento", "tamaño", "frutos") | Para cambiar simultáneamente el nombre de las columnas del objeto mibase. OJO: usar el mismo número de nombres. |
| as... | |
| mibase$trat <- as.numeric(mibase$trat) | Para forzar a una columna a ser numérica |
| mibase$trat <- as.factor(mibase$trat) | Para forzar a una columna a ser factor. Extremadamente útil si hemos codificado los niveles de un factor utilizando números. |
| factor | |
| mibase$pob <- factor(mibase$pob, levels = c("b", "c", "a")) | Función más flexible que as.factor. levels ayuda a determinar cuál de los niveles es tomado como referencia (el primero de la lista). Esto es útil para realizar comparaciones. |
| mibase$pob <- factor(mibase$pob, levels = c("b", "c", "a"), ordered = TRUE) | Indica que los niveles de una factor poseen un orden natural (de menor a mayor), por ejemplo el orden de llegada en una carrera. Útil en ciertos análisis. |
| mibase$pob <- factor(mibase$pob, levels = c("b", "c", "a"), labels = c("Bolivia", "Colombia", "Argentina")) | Permite renombrar los niveles de un factor. Seguir el orden definido por levels, remplazando valores |
| mibase$frutos[1] <- 3 | Remplaza el primer valor (10) de la columna frutos con el valor 3. Si quisieras remplazar el segundo valor seria (mibase$frutso[2] <- 3) |
| mibase$frutos[which(mibase$frutos == 10)] <- 3 | Remplaza todos los valores que eran 10 por el valor 3. Se pueden utilizar < (menor),> (mayor),!= (distinto) |
Trabajando con filas[editar]
Las actividades más comunes con filas son quitar una fila, cambiarla de lugar, o cambiar de nombre. Ocasionalmente, algunos sets de datos requieren tener filas con nombres. Por ejemplo si tenemos una matriz de presencia/ausencia de especies (filas) en diferentes sitios (columnas) suele ser útil indicarle a R que coloque los nombres de las especies como nombres de filas.
| rownames | |
|---|---|
| nombres<-c("a", "b", "c", "d", "e", "f", "g", "h", "i", "j", "k", "l", "m", "n","o", "p", "q", "r","s", "t", "u", "v", "w", "x")
rownames(mibase) <- nombres |
Para asignarle por nombre una letra a cada fila |
| Creando nueva base | |
| mibase.2 <- mibase[-c(3, 5), ] | Creamos una nueva base (mibase.2) igual a la anterior, pero sin las filas 3 y 5. OJO: es conveniente no utilizar el mismo nombre, por las dudas nos arrepintamos de esta acción. |
Uniendo bases de datos[editar]
Hay dos formas básicas en las que se pueden unir bases de datos: horizontalmente o verticalmente, es decir, o se adhieren las columnas de ambas bases o las filas.
- Adhesión de columnas (horizontal): se debe estar seguro que ambas bases de datos tengan el mismo número de filas.
- Adhesión de filas (vertical): se debe estar seguro de tener el mismo número de columnas y que el nombre de las columnas sea igual.
Para este ejemplo vamos a utilizar dos bases de datos nuevas.
- mibase2 contiene una columna llamada largo con datos numéricos y una columna llamada trat2 que consiste en un factor con dos niveles: "a" y "b".
- mibase3 contiene las mismas variables que la base original, pero agrega los datos de una cuarta población.
mibase2
| obs | largo | trat2 |
|---|---|---|
| 1 | 4.726665 | a |
| 2 | 3.263297 | a |
| 3 | 3.411479 | b |
| 4 | 3.991426 | b |
| 5 | 4.066395 | a |
| 6 | 3.762102 | a |
| 7 | 4.39818 | b |
| 8 | NA | b |
| 9 | 3.712131 | a |
| 10 | 4.078987 | a |
| 11 | 3.994718 | b |
| 12 | 3.992025 | b |
| 13 | 4.756946 | a |
| 14 | 3.64809 | a |
| 15 | 4.05796 | b |
| 16 | 3.931277 | b |
| 17 | 3.578331 | a |
| 18 | 3.673713 | a |
| 19 | 3.785033 | b |
| 20 | 5.031436 | b |
| 21 | 2.929234 | a |
| 22 | 2.965337 | a |
| 23 | 4.543052 | b |
| 24 | 3.399721 | b |
mibase3
| obs | pob | trat | tam | frutos |
|---|---|---|---|---|
| 25 | d | 1 | 1.456063 | 0 |
| 26 | d | 1 | 1.312139 | 2 |
| 27 | d | 1 | 1.792546 | 5 |
| 28 | d | 1 | 1.809306 | 3 |
| 29 | d | 2 | 1.570933 | 1 |
| 30 | d | 2 | 1.084462 | 5 |
| 31 | d | 2 | 1.439948 | 10 |
| 32 | d | 2 | 1.773188 | 6 |
| cbind | |
|---|---|
|
BASE.A <- cbind(mibase, mibase2) |
Une las columnas de ambas bases (c=columna, bind= unión). Usar cbind sólo si mibase y mibase2 son dos data.frame o dos vectores de un mismo tipo (por ej. dos columnas numéricas o dos columnas de caracteres). Ambas bases deben tener el mismo número de filas (o el mismo número de elementos si son vectores). |
| rbind | |
|
BASE.B <- rbind(mibase, mibase3) |
Une las filas de ambas bases (r=row, bind= unión). Ambas bases deben tener el mismo número de columnas. |
| data.frame | |
|
BASE.A <- data.frame(mibase, mibase2) n.base <- data.frame(tratamiento = mibase$trat, largo = mibease2$largo) |
Une las bases de datos por columnas. A diferencia de cbind, puede usarse para construir data.frames a partir de vectores (columnas) de distinto tipo y también se puede asignarles un nuevo nombre. |
| merge | |
|
# versión reducida de mibase2 M2 <- mibase2[c(1:5,7:22),] BASE.C <- merge(mibase, M2, by = "obs", all = TRUE) |
Útil cuando deben coincidir dos bases de datos con distinto número de filas (siempre que tengamos una columna indicando a qué observación corresponde, en este caso es obs). Los espacios faltantes son llenados con NA. |
| union, intersect, setdiff | |
|
union(mibase$pob, mibase2$trat) |
Define los elementos presentes en al menos un vector. |
|
intersect(mibase$pob, mibase2$trat) |
Define los elementos que se comparten entre vectores. |
|
setdiff(mibase$pob, mibase2$trat) |
Define los elementos que difieren entre vectores. |
| Tips: para situaciones más complicadas.
Para casos más complicados usa el paquete multitable que permite unir muchas bases con diferentes dimensiones (i.e. diferente número de columnas y filas) y te ahorra muchos problemas. Por ejemplo, si quisieras integrar la información de temperatura y precipitación de cada población a cada una de las observaciones realizadas, sólo tendrías un valor climatológico por población mientras que cada población tendría ocho observaciones. Claramente las dimensiones entre estas bases serían diferentes. |
Dividiendo bases de datos[editar]
La división de las bases de datos se realiza fácilmente utilizando la función subset. Esta función tiene dos argumentos principales: el objeto a ser dividido y una condición lógica que explica cómo realizar la división. Sólo es necesario tener en cuenta el uso símbolos para comparar y de algunos operadores lógicos.
| subset | |
|---|---|
|
base1 <- subset(mibase, mibase$pob == "a") |
División de acuerdo a un factor. Se crea una nueva base de datos (base1) que almacena aquella parte de la base original (mibase) donde la columna pob indique que esos datos pertenecen a la población "a". |
|
base2 <- subset(mibase, mibase$pob == "a" & mibase$pob == b) base2 <- subset(mibase, mibase$pob %in% c ("a","b")) |
Permite armar una nueva base que incluye a la poblacion "a" y "b".
La rutina de abajo es un sinónimo pero con la ventaja de que permite incluir muchas poblaciones (niveles) de manera mas eficiente, incluyéndolas en un vector c("a","b", ...etc) |
|
base2 <- subset(mibase, mibase$pob != "a") |
División de acuerdo a un factor. La nueva base almacena los datos de la base original pero excluyendo los datos de la población "a". |
|
base3 <- subset(mibase, mibase$tam <= 5) |
División de acuerdo a una variable numérica. La nueva base almacena los valores que cumplan con la condición de que el tamaño sea menor o igual (<=) a 5. |
|
base4 <- subset(mibase, mibase$tam > 3 | mibase$pob == "c") |
Combinación de casos anteriores. La nueva base almacena los valores que cumplan con la condición de que el tamaño sea mayor que 3 o (|) que pertenezca a la población "c". |
Tips: Recordatorio de símbolos.
|
Ordenando la base de datos[editar]
Muchas veces queremos visualizar la base de datos con cierto orden. He aquí cómo hacerlo.
| Ordenando columnas [,] | |
|---|---|
|
baseCol <- mibase[, c(5, 4, 3, 2, 1)] |
Ordenando columnas. Invierte el orden de las columnas. La última columna es la primera ahora. De esta forma se pueden diseñar diferentes disposiciones de las columnas, eliminar alguna de ellas (dejar fuera por ejemplo la columna 1), etc. |
| Ordenando filas (order) | |
|
mibase.o <- mibase[order(mibase$tam), ] mibase.o <- mibase[order(mibase$tam, decreasing = TRUE), ] |
Ordena en forma ascendente la base en función de la columna indicada en order (tam en este caso). La linea de abajo hace lo mismo pero en forma descendente. |
|
baseFila3 <- mibase[order(mibase$trat, mibase$tam), ] |
Ordenando la base en función de arreglar dos columnas (pob y tam) de manera ascendente. |
|
baseFila4 <- mibase[order(mibase$trat, -(mibase$tam), ] |
Ordenando la base en función de arreglar dos columnas: trat de manera ascendente y tam de manera descendente |
| Ordenando en forma aleatoria (sample) | |
|
baseFila6 <- mibase[sample(1:nrow(mibase), replace = FALSE), ] |
Ordenar de manera aleatoria las filas de la base |
|
baseFila7 <- mibase[sample(1:nrow(mibase), replace = TRUE), ] |
Ordena de manera aleatoria las filas de la base pero con remplazo, es decir algunas filas podrían aparecer repetidas y otras no aparecer en absoluto. La base del bootstrap. |
ESTADÍSTICA DESCRIPTIVA Y PRUEBAS BÁSICAS[editar]
Las funciones básicas para extraer información de parámetros como media y varianza son sencillas y similares. Como ayuda general, si se aplican sobre datos que contienen NA el resultado será NA también, por lo cual hay indicarle a las funciones que los elimine.
Medias, varianzas y demás[editar]
| mean | |
|---|---|
|
M <- mean(mibase$tam, na.rm = TRUE) |
Calcula la media. El primer argumento es el nombre de la columna, el segundo indica qué hacer con los NA (en este caso removerlos, lo más aconsejable) |
| median | |
|
Me <- median(mibase$tam, na.rm = TRUE) |
Calcula la mediana. Mismos argumentos que la media. |
| var | |
|
V <- var(mibase$frutos, na.rm = TRUE) |
Calcula la varianza muestral (sobre N-1). Mismos argumentos que la media. |
| sd | |
|
SD <- sd(mibase$frutos, na.rm = TRUE) |
Calcula la desviación estándar muestral (sobre N-1). Mismos argumentos que la media. |
| quantile | |
|
Qu <- quantile(mibase$tam, probs = seq(0, 1, by = 0.25), na.rm = TRUE, type= 7) |
Calucula los cuantiles. El argumento probs determina cuantos cuantiles calcular, por ej. seq (0, 1, 0.25) indica que calculará el mínimo (0), los cuantiles 25, 50 y 75% y el máximo (1). na.rm indica que hacer con los NA. type = 7 se debe a que, curiosamente, existen nueve formas de calcular cuantiles. |
| range | |
|
R <- range(mibase$frutos, na.rm = TRUE) |
Muestra el valor mínimo y máximo. |
| summary | |
|
summary(mibase) |
Obtienes la media, medianas, quantiles, valores mínimos y máximos de aquellas columnas que sean numéricas. Para aquellas que son factores te indica cuantos niveles tiene el factor y el numero de replicas que tiene cada nivel (i.e. a = 8 observaciones, b = 8 observaciones, etc.) |
Funciones que retornan matrices[editar]
Las matrices de varianza-covarianza y de correlación de un set de datos pueden obtenerse con las funciones var y cor, respectivamente. Presta atención a la forma de evitar los NA de la última.
| var | |
|---|---|
|
VC <- var(mibase[, 4:5], na.rm = TRUE) |
Calcula un objeto matrix que posee las varianzas muestrales en su diagonal y las covarianzas en el resto de las celdas. |
| cor | |
|
CO <- cor(mibase[, 4:5], use = "complete.obs") CO <- cor(mibase[, 1:6], use = "complete.obs", method = "spearman") |
Calcula la matriz de correlación. En el caso de "complete.obs" eliminará todas las filas del set de datos que tengan un NA. Se puede cambiar el método para estimar la correlación agregando por ejemplo: method = "spearman" |
Pruebas básicas[editar]
| t.test | |
|---|---|
|
#paso previo t1 <- subset(mibase, mibase$trat == "1") t2 <- subset(mibase, mibase$trat == "2") #prueba t t.test(t1$tam,t2$tam, na.action = na.omit) |
Prueba t para dos variables continuas. Para hacer una prueba t pareada agrega: paired = TRUE. |
|
t.test(tam ~ trat, data = mibase, na.action = na.omit) |
Una variable continua y una variable binaria (por ej. Trat 1 vs. Trat 2) |
|
t.test(mibase$tam, mu = 5, na.action = na.omit) |
Prueba t para una media, requiere una variable continua y un valor de media |
| wilcox.test | |
|
wilcox.test(tam ~ trat, data = mibase, na.action = na.omit) |
La versión no paramétrica de la prueba t. Tiene las mismas opciones que t.test. |
| cor.test | |
|
cor.test(mibase$frutos, mibase$tam, use = "complete.obs", method = "pearson") |
Estima la correlación entre ambas variables y su nivel de significancia. El método de estimación puede ser cambiado entre "pearson", "kendall" o "spearman". |
| chisq.test | |
|
ji <- matrix (c(9,4,3,9),2) #matriz prueba.ji<- chisq.test(ji) prueba.ji |
Primero tenemos que hacer una matriz que contenga una tabla de contingencia. |
| fisher.test | |
|
prueba.F <- fisher.test(ji) prueba.F |
Se usa cuando el 75% de las entradas de la tabla de contingencia tienen valores menores a 5. |
| kruskal.test | |
|
kruskal.test(frutos ~ trat, data = mibase) |
Prueba no paramétrica equivalente a un ANOVA de una via. |
GRÁFICOS BÁSICOS[editar]
R tiene gran poder para hacer gráficos, no lo desperdicies. R tiene diferentes paquetes para graficar, entre ellos destacamos:
- graphics Contiene las funciones básicas para graficar. Hace gráficos poco ostentosos pero que en muchas ocaciones son perfectos para publicaciones en revistas indexadas.
- lattice y grid Contienen gráficos útiles para datos multivariados y explorar interacciones.
- ggplot2 Sirve para hacer gráficos modernos y elegantes.
Otra cosa importante a considerar es como se construyen los objetos gráficos. En general tiene tres niveles de construcción:
- Funciones gráficas de alto nivel Crean un gráfico que es dibujado en una ventana gráfica. A este nivel, no solo se crea el gráfico en bruto sino que se le puede hacer ciertas modificaciones como cambio de color, tamaño, tipo de fuente y otras características.
- Funciones gráficas de bajo nivel Agregan más información a un gráfico ya existente, como puntos extras, líneas, funciones y leyendas.
Funciones básicas de alto nivel[editar]
| plot | |
|---|---|
|
plot(-1:1, -1:1, type = "n", xlab = "eje x", ylab = "eje y") |
Abre un espacio gráfico con una escala definida (-1:1) en cada uno de sus ejes, pero no incluye ningÚn dato. Con type = "n" (nulo) no se grafica punto alguno. |
|
plot(mibase$tam) |
Grafica los valores de x (en el eje y) ordenados en el eje x |
|
plot(mibase$tam, mibase$frutos) plot (mibase$frutos ~ mibase$tam) plot (mibase$pob, mibase$tam) |
Grafico bivariado de x (en el eje x) e y (en el eje y) (plot(x, y)). Si x es un factor R hará un boxplot |
| boxplot | |
|
boxplot(mibase$tam) |
Grafico de caja y bigotes (box and whiskers). |
| coplot | |
|
coplot (frutos ~ tam|pob, data = mibase) |
Grafico bivariado entre y ~ x donde dicha relación está condicionada por una tercera variable, en este caso pob. Es un gráfico feo. |
| interaction.plot | |
|
interaction.plot(x.factor = mibase$pob, trace.factor = mibase$trat, response = mibase$frutos) |
Gráficos para ver interacciones en modelos lineales. Influencia de dos factores (pob y trat) sobre una variable de respuesta númerica (frutos). El factor x es el que ocupa el eje x. El factor "trace" se representa con lineas de diferente diseño. |
| pairs | |
|
pairs (mibase) |
Dibuja todas las posibles gráficas bivariadas entre todas las columnas |
| pie | |
|
pie(c(55, 32, 12)) |
Hace gráfico de pastel donde divide el pie en función de los nÚmeros introducidos en un vector. |
| hist | |
|
hist(mibase$tam) hist (mibase$tam, breaks = 10) hist (mibase$tam, freq = FALSE) points(density(mibase$tam, na.rm = TRUE) |
Grafica un histograma de frecuencias de x. Incluir “breaks” permite manipular el número de columnas. Incluir freq = FALSE convierte al eje de las y en densidad, es decir, el histograma tiene un área de 1. La linea points sobrelapa, sobre el histograma, un gráfico de densidad. |
| qqnorm | |
|
qqnorm(mibase$tam) |
Cuantiles de x con respecto a los esperado bajo una distribución normal |
| qqplot | |
|
qqplot (mibase$frutos, mibase$tam) |
Cuantiles de y con respecto a los cuantiles de x (qqplot(x,y)) |
| library(sciplot); bargraph.CI | |
|
library(sciplot) bargraph.CI(x.factor = pob, response = frutos, data = mibase) bargraph.CI(x.factor = pob, response = frutos, group = trat, data = mibase) |
Gráfico de barras usando errores estándar. La otra linea divide las columnas por el grupo “trat” |
| library(sciplot); lineplot.CI | |
|
library(sciplot) lineplot.CI(x.factor = pob, response = frutos, group = trat, data = mibase) |
Gráfico de lineas incluyendo errores estándar. |
Funciones básicas de bajo nivel[editar]
Para poder visualizar el efecto de estas funciones es necesario ya contar con un gráfico, por ejemplo plot(mibase$tam, mibase$frutos)
| points | |
|---|---|
|
points(mibase$tam, mibase$frutos, pch = 3) |
Agrega puntos al gráfico. En este caso para que se note utilizamos pch para introducir un formato de punto diferente. Ver abajo los tipos de puntos disponibles. |
| lines | |
|
lines(mibase$tam, mibase$frutos) |
Agrega lineas al gráfico. |
| text | |
|
text(5, 12, "hola") |
Agrega el texto "hola" en la coordenada x = 5 e y = 12. |
| segments | |
|
segments(3, 4, 4, 5) |
Agrega segmentos (lineas) desde la coordenada x0; y0 hasta x1; y1. Notar que las “x” e “y” pueden ser vectores de igual longitud |
| arrows | |
|
arrows(3, 4, 4, 5) |
Agrega flechas de la misma forma que se agregan los segmentos. |
| abline | |
|
abline(a = 0, b = 1) |
Agrega una línea con ordenada al origen = a y pendiente = b. |
|
abline(fit) |
Agrega la recta de regresión obtenida a partir de realizar modelo lineal (objeto de nombre fit) |
|
abline(h = 4) |
Agrega una línea horizontal en la ordenada = y. |
|
abline(v = 4) |
Agrega una línea vertical en la abscisa = x. |
| polygon | |
|
polygon(c(2, 4, 6, 4, 2), c(8, 4, 8, 12, 8)) |
Para dibujar un polígono se necesita usar vectores que indican las coordenadas de las sucesivas esquinas. |
| axis | |
|
axis(side = 3) |
Agrega ejes al grafico: abajo (side = 1), a la izquierda (side = 2), arriba (side = 3) y derecha (side = 4). |
| title | |
|
title("hola") |
Agrega un título. |
Argumentos comunes a varias funciones gráficas[editar]
Todos estos argumentos pueden ser incluidos dentro de una función de alto nivel. Por ejemplo: plot (mibase$tam, mibase$frutos, axes = FALSE, bty = "red", col = "black", etc)
| axes = | TRUE o FALSE, dibuja o no los ejes. |
|
bty = |
Tipo de caja: "o", "I", "7", "c", "u", "ou", "n" (n no dibuja la caja) |
|
col = |
Controla el color de los símbolos o líneas (ver tabla de referencia de colores). |
|
type = |
"p" puntos (por defecto), "l" líneas, "b" puntos conectados por líneas, "o" líneas sobre los puntos, "h" líneas verticales, "s" en escalera (un paso horizontal, uno vertical), "S" en esacalera (un paso vertical, uno horizontal). |
|
xlim =, ylim =, zlim = |
Define los límites de los ejes (ej. xlim = c(0, 12), o sea que el eje x vaya de la coordenada 0 a la 12) |
|
xlab =, ylab =, zlab = |
Para establecer la leyenda de los ejes (ej. xlab = "número de ranas") |
|
main = |
Para el titulo principal (ej. main = "Gráfica principal") |
|
lwd = |
Controla el grosor de las líneas (por ej. lwd = 1 para tamaño normal, lwd = 2 es el doble de lo normal, lwd = 0.5 es la mitad de lo normal). |
|
lty = |
Controla el tipo de línea: sólidas (lty = 1), con guiones (lty = 2), con puntos (lty = 3), puntos y líneas (lty = 4), etc. |
|
pch = |
Controla el tipo de símbolo, por ejemplo cambia de puntos a cruces (ver tabla de referencia de diseño de símbolo). Además de los símbolos de la tabla puede utilizarse cualquier símbolo del teclado poniéndolo entre comillas. Por ejemplo pch = "j". |
|
font = |
Controla el tipo de fuente: 1 normal, 2 itálica, 3 negrita, 4 negrita itálica. |
|
ps = |
Controla el tamaño de textos y símbolos. 1 es el tamaño normal, 3 es el triple de lo normal, 0.5 es la mitad de lo normal, etc. |
|
bg = |
Controla el color de fondo. |
|
cex = |
Define el tamaño relativo de los símbolos (cex = 2) y letras (ejes y títulos), para esto último existen cex especiales: cex.axis, cex.lab, cex.main, cex.sub. |
Símbolos para graficar[editar]
Colores para graficar[editar]
Tendrás que hacer mucho zoom para leer los nombres de los colores. Si prefieres puedes construir tu propia tabla de colores y guardarla en con los siguientes comandos:
| Tips: rutina para construir una tabla de colores en pdf.
setwd("C:/Users/Yo/Mis Documentos") # recuerda cambiar el directorio A <- rep(seq(1, 16, by = 2), 30) B <- sort(rep(seq(1, 30), 8), decreasing = TRUE) pdf(file = "hoja colores 1 de 3.pdf", width = 8.5, height = 11, paper="a4") par(mar = c(0.1, 0.1, 2, 0.1)) plot(A, B, axes = FALSE, pch = 15, cex = 2.8, col = colors()[1:240], xlim = c(0, 17)) text(A+0.27, B, colors()[1:240], cex = 0.45, adj = 0) title(main = list("COLORES EN R - HOJA 1 \n por Santiago Benitez-Vieyra", cex = 0.8)) dev.off()
par(mar = c(0.1, 0.1, 2, 0.1)) plot(A, B, axes = FALSE, pch = 15, cex = 2.8, col = colors()[241:480], xlim = c(0, 17)) text(A+0.27, B, colors()[241:480], cex = 0.45, adj = 0) title(main = list("COLORES EN R - HOJA 2 \n por Santiago Benitez-Vieyra", cex = 0.8)) dev.off()
par(mar = c(0.1, 0.1, 2, 0.1)) plot(A, B, axes = FALSE, pch = 15, cex = 2.8, col = c(colors()[481:657], rep("white", 63)), xlim = c(0, 17)) text(A+0.27, B, c(colors()[481:657], rep("", 63)), cex = 0.45, adj = 0) title(main = list("COLORES EN R - HOJA 3 \n por Santiago Benitez-Vieyra", cex = 0.8)) dev.off() |
Sobre el espacio de graficación[editar]
R tiene un espacio de graficación el cual puede ser subdividio para graficar más de un gráfico por ventana. Para esto se corta el espacio de graficación siguiendo la logica de una matriz (es decir, se cuadricula el espacio en filas y columnas).
| layout | |
|---|---|
|
layout(matrix(1:4, 2, 2)) hist (mibase$tam, main = "histograma tamaño") hist (mibase$frutos, main = "histograma frutos") plot (mibase$pob, mibase$tam, main = "tam x pob") plot (mibase$pob, mibase$frutos, main = "frutos x pob") layout (1) |
Subdivide la ventana en 4 ventanas. Dos filas y dos columnas. En este ejemplo se hacen 4 gráficos. layout (1) regresa al espacio original el cual no esta subdividido. |
|
layout(matrix(1:4, 2, 2) , widths = c(1, 2), heights = c(2, 1)) layout.show(4) layout(1) |
Para modificar el tamaño relativo de los gráficos. layout.show permite ver como se dividen los marcos de graficación antes de realizar los gráfios. |
| par | |
|
par(mfrow = c(2, 2)) hist(mibase$tam, main = "histograma tamaño") hist(mibase$frutos, main = "histograma frutos") plot(mibase$pob, mibase$tam, main = "tam x pob") plot(mibase$pob, mibase$frutos, main = "frutos x pob") par () |
Otra forma de dividir el espacio es usar esta función. |
|
par(mfcol = c (1, 2)) hist (mibase$tam, main = "histograma tamaño") hist (mibase$frutos, main = "histograma frutos") par() |
En este caso cambiamos mfrow por mcol que altera tambien el espacio. |
Guardando gráficos[editar]
Hay varios formatos en los que se pueden guardar las figuras. Para este ejemplo usaremos el histograma de la variable tamaño (tam). Se puede establecer previamente el directorio donde se van a guardar los gráficos usando setwd(). La forma más práctica de guardar los gráficos depende del sistema operativo que tengas y de qué programa pienses usar para abrirlos o editarlos. En windows lo mas sencillo es seleccionar el gráfico con el puntero del mouse y dar click en el botón derecho y salvar el formato que se requiera. La ventaja de usar las funciones que mostramos a continuación es que se puede definir desde R el tamaño final que se quiere para la figura, además de automatizar el proceso. Como regla general, todo el proceso de guardar un gráfico tiene tres pasos: la primera función (svg, pdf, jpeg, etc.) crea el archivo. En el segundo paso ingresamos todos los comandos gráficos necesarios, que "rellenan" el archivo. Finalmente dev.off() hace que R cierre el archivo de modo que se pueda abrir con otros programas.
| jpeg | |
|---|---|
|
jpeg(file = "mi_grafico.jpg", width = 12, height = 10, units = "cm", quality = 95, res = 300) hist (mibase$tam, main="histograma tamaño") dev.off() |
Para generar una figura en jpeg. width y height determina el ancho y alto del objeto. units determina las unidades (pixeles por defecto) en las que será guardado. quality determina el nivel de no-compresión y res el nivel de resolución. Este es el único formato que permite manipular las unidades de medida.
dev.off () es fundamental para poder abrir el archivo, si no ejecutamos ese comando el archivo con el gráfico permanecerá "ocupado" por R. |
|
pdf(file = "mi_grafico.pdf", width = 7, height = 6, paper = "a4") hist (mibase$tam, main="histograma tamaño") dev.off() |
Para generar una figura en pdf. paper determina el tamaño del papel. El tamaño por defecto es igual al gráfico.width y height se encuentran en pulgadas (1 in = 2.54 cm). |
| eps | |
|
postscript(file = "mi_grafico.eps", width = 7, height = 6, paper = "a4", horiz = TRUE) hist (mibase$tam, main="histograma tamaño") dev.off() |
Para generar una figura eps. horizontal (FALSE) o vertical (TRUE) define la orientación de la figura. |
| svg | |
|
svg(file = "mi_grafico.svg", width = 7, height = 6) hist (mibase$tam, main="histograma tamaño") dev.off() |
Para generar figuras svg. svg (Scalable vector graphics) es el formato vectorial libre y puede ser abierto y editado por softwares libres (nuestros preferidos) como Inkscape. Puedes usar este último programa para pasar del formato svg a otros como eps o pdf. |
MODELOS LINEALES: REGRESIÓN, ANOVA Y DEMÁS[editar]
En esta sección mostramos la sintaxis para realizar diferentes pruebas estadísticas. No pretendemos explicar los fundamentos estadísticos de cada prueba, para eso están los libros y cursos de estadística.
Exploración visual de datos[editar]
Los gráficos son una gran herramienta para conocer nuestros datos. La visualización de los datos es fundamental tanto para confirmar que nuestra base de datos está libre de errores de transcripción o de toma de datos, como para entender la naturaleza de la información que colectamos (i.e. nuestros datos). Grandes errores pueden ser detectados de esta manera. Repetiremos algunas funciones que ya se vieron previamente pero con la intención de mostrar explícitamente las herramientas básicas para esta exploración.
| Detección de outliers | |
|---|---|
|
plot(mibase$frutos, pch = ".") text(mibase$frutos,labels = rownames(mibase), cex = 0.8) |
Cualquier punto muy alejado de la nube será potencialmente un outlier. La función de bajo nivel text te puede ayudar a identificar cual es la fila que se tiene que remover para quitar el outlier. En este caso se puede notar que la fila 23 esta un poco alejada de la nube principal. No remuevas los outliers solamente porque "te parece" o "me hacen salir mal el análisis". ¡Eso es trampa! Remueve con seguridad los outliers que provengan de datos mal tomados o mal escritos, pero si el outlier es "natural" no deberías removerlo, o al menos deberías reportar tu análisis por duplicado, con y sin esa observación. |
|
boxplot(mibase$frutos) |
El boxplot nos confirman el outlier. |
| Explorar la distribución de la variable | |
|
hist(mibase$frutos) d <- density(mibase$frutos) plot(d) |
Puede usarse el histograma o generar un gráfico de densidad. |
| Relación entre variables | |
|
pairs(mibase) |
Para ver la relación entre todo el conjunto de variables consideradas. |
|
plot(mibase$tam, mibase$frutos) text(mibase$tam, mibase$frutos, labels = rownames(mibase), cex = 0.8) |
Para ver cada par de variables |
Construcción de modelos lineales y resúmenes básicos[editar]
Los modelos lineales tienen la ventaja de poder de poder incrementar en nivel de complejidad dependiendo de las necesidades y el diseño experimental utilizado. Los resultados de estos modelos pueden resumirse de dos maneras básicas, usando: summary y anova.
- summary da la tabla de coeficientes. En el caso de una regresión los coeficientes son las conocidas ordenada al origen (β0) y la pendiente (β1) o pendientes parciales (β1, β2, β3) cuando el modelo contienen mas de un variable independiente. Para cada coeficiente hay una prueba t asociada que indica si difiere de 0. Cuando el modelo es una análisis de la varianza los coeficientes corresponden a las diferencias de medias entre los niveles de un factor y uno tomado como referencia alfabéticamente. Esta función también da los parámetros generales del modelo tales como el coeficiente de determinación (R2), el valor de F, los grados de libertad y el valor de P.
- anova da la tabla de análisis de varianza que muestra para cada variable los grados de libertad, la suma de cuadrados, los cuadrados medios, una prueba F con su correspondiente valor de P que indica si la porción de la variabilidad explicada por esa variable es significativa en relación con la variabilidad del error. Por defecto la función anova utiliza una suma de cuadrados de tipo I (secuencial). Para las otros tipos de sumas de cuadrados (II y III) utilizar la función Anova del paquete car. Por ejemplo Anova(nombre_modelo, type ="II")
| lm | |
|---|---|
|
fit <- lm(frutos ~ tam, data = mibase) summary(fit) anova(fit) |
Regresión lineal simple. La variable tam es continua. |
|
fit <- lm(frutos ~ pob, data = mibase) summary(fit) anova(fit) |
Análisis de la varianza (ANOVA) de una vía. La variable pob es categórica con tres niveles. Si una variable es categórica y esta codificada con números (ej. la variable trat) es necesario transformarla previamente utilizando factor o as.factor. |
|
fit <- lm(frutos ~ tam + pob, data = mibase) summary(fit) anova(fit) |
Análisis de la covarianza (ANCOVA). Dos variables independientes, una continua y otra categórica. Para ver modelos más complejos ver tabla de fórmulas. |
| aov | |
|
fit <- aov(frutos ~ pob, data = mibase) summary(fit) TukeyHSD(fit) |
Forma alternativa de hacer un análisis de varianza. En este caso el summary da la tabla de análisis de la varianza y no da la tabla de coeficientes. De aqui se puede obtener la prueba de diferencia de medias de Tukey (utilizando la función TukeyHSD) |
Las formulas en los modelos lineales pueden representar distintas combinaciones de variables para ajustar modelos aditivos, con interacción (no aditivos), polinomios y modelos anidados.
| Y ~ 1 | Modelo sin efecto. Solo intercepto |
|
Y ~ A |
Modelo lineal simple (regresión lineal simple o ANOVA de una via) |
|
Y ~ A - 1 |
Modelo lineal simple cuyo intercepto esta forzado a ser cero. |
|
Y ~ A + B |
Modelo multiple aditivo (regresión multiple, ANCOVA o ANOVA de dos vias) |
|
Y ~ A + B + A:B Y ~ A * B Y ~ (A + B)^2 Y~ (A + B + C)^3 |
Modelo con interacción (A:B) y diferentes formas sinónimas de escribirlos, que son más eficientes cuando hay muchas variables. La ultima forma implica un modelo donde se ajustan todos los efectos principales (A, B, C), todas las interacciones dobles (A:B, A:C, B:C) y la interacción triple (A:B:C). Es posible escribir las interacciones hasta cualquier grado deseado. |
|
Y ~ A + B %in% A Y ~ A/B |
Modelo con efecto principal de A y efecto de B anidado en A (dos formas sinónimas de escribirlo). |
|
Y~ A + I(A^2) |
Modelo con efectos lineales y cuadráticos. I permite introducir efectos no lineales (cuadráticos, cúbicos, logarítmicos etc). |
Salida de un modelo lineal[editar]
Los objetos creado a partir de un modelo lineal (en este caso el objeto fit) contienen mucha mas información que la que muestran summary y anova. Este tipo de información puede usarse para pruebas subsecuentes y análisis más detallados.
| coefficients | |
|---|---|
|
coef.fit <- coefficients(fit) coef.fit <- fit$coef |
Permite extraer los coeficientes. Hay dos formas alternativas de hacerlo. Guardamos los valores en un objeto al que llamamos coef.fit. |
| confint | |
|
confint(fit, level = 0.95) |
Genera los intervalos de confianza (inferior y superior) de los coeficientes estimados. El nivel de confianza puede manipularse con level. |
| residuals | |
|
res.fit <- residuals(fit) res.fit <- fit$resid |
Permite extraer los residuales del modelo. Es importante guardarlos en un nuevo objeto (que llamamos res.fit) para después poder utilizarlos. |
| fitted | |
|
fit.fit <- fitted(fit) fit.fit <- fit$fit |
Permite extraer los valores predichos del modelo. Igual que arriba es importante guardarlos en un objeto (fit.fit). |
Cuando el problema no es obtener los valores predichos del modelo sino obtener predicciones para una nueva serie de valores podemos usar la función predict.
| predict | |
|---|---|
|
x <- seq(1, 5, 0.1) fit <- lm(frutos ~ tam, data = mibase) y <- predict(fit, data.frame(tam = x), se.fit = TRUE) plot(x, y$fit, type = "l") points(x, y$fit + y$se.fit) points(x, y$fit - y$se.fit) |
Primero se genera un nuevo grupo de valores para la variable independiente (x). seq crea una secuencia del 1 al 5 en intervalos de 0.1.
recordando el modelo... Usamos la función predict a partir del objeto fit y de los valores de x recién creados. Predice nuevos valores de frutos basados en el ajuste entre frutos y tamaño pero ahora utilizando los valores de x. Si se le añade el se.fit = TRUE generará una columna de predichos y otra de errores estándar. Toda esa información se puede graficar. |
Diagnósticos de modelos[editar]
La violación del supuesto de no normalidad afectará la significancia de los terminos evaluados. Por otro lado, la presencia de observaciones influyentes afectará la estimación de los parámetros. Esto no quiere decir necesariamente que hay remover esas observaciones. Generalmente, lo que indica es que el muestreo fue deficiente o que hubo un error en la toma de datos. Hay dos formas de analizar los datos: la gráfica y la numérica. Te recomendamos confiar en la gráfica (aunque requiere práctica) porque cuando el número de observaciones es muy pequeño los diagnósticos numéricos son poco sensibles y, cuando es muy grande, son excesivamente sensibles.
Diagnósticos Gráficos[editar]
| plot | |
|---|---|
|
layout(matrix(1:4, 2, 2)) plot(fit) layout(1) |
La función de plot aplicada sobre un objeto "modelo lineal" (fit) arroja 4 gráficos de diagnostico. En todos los gráficos las observaciones que podrían generar algún problema están señalizadas con un número que corresponde al número de la fila. |
- Gráfico de residuales vs. predichos. Para probar linealidad y homogeneidad de residuos (homocedasticidad). La linealidad se examina evaluando que la linea roja sea lo mas horizontal posible y sin curvatura. Si tuviera curvatura indicaría que el modelo puede necesitar un termino de ajuste no lineal (i.e. cuadrático, logarítmico etc) o que hay una variable importante no incluida en el modelo. La homogeneidad de varianza se examina observando que la distribución de los valores (puntos) sea lo más homogénea posible en el espacio ("como una nube"). Cuando esto no se cumple el patrón más típico es de forma de trompeta, lo que indica que valores más altos son también más variables.
- Raíz cuadrada de residuales estandarizados (i.e. residuales studentizados) vs. predichos. Prueba homogeneidad de residuos (homocedasticidad). Si la linea de roja muestra una tendencia de los datos es también falta de homogeneidad. Al estar estandarizados, los valores por arriba y por abajo de 2 son observaciones con residuos extremos lo que implica que el modelo no los ajusta bien (su valor predicho por el modelo y su valor observado están muy alejados).
- Normal Q-Q plot. Este gráfico prueba la normalidad de los residuos. Los valores (puntos) deben estar lo más cercanos de la linea diagonal. Las desviaciones indican desajuste.
- Residuales estandarizados vs leverage (gráfico de influencias). Un observación será muy influyente si posee un valor residual muy grande y al mismo tiempo un leverage muy alto (esto último indica que el valor de la variable independiente para esa observación es muy grande o muy pequeño). Esto significa que la presencia o ausencia de ese punto podrá afectar los resultados y párametros del modelo. Una medida de esa influencia es la distancia de Cook (que combina residuales y leverage) la cual aparece dibujada en el gráfico como curvas. Por arriba de 1 se considera influyente, aunque no hay una regla estricta.
Diagnósticos numéricos[editar]
| shapiro.test | |
|---|---|
|
fit <- lm(frutos ~ tam + pob, data = mibase) res.fit <-residuals(fit) shapiro.test(res.fit) |
Prueba la normalidad de los residuos. Para eso primero guardamos los residuos de nuestro modelo en el objeto res.fit. Un resultado significativo (P < 0.05) indica que la distribución de los residuales es significativamente diferente a la esperada para una distribución normal. |
| barlett.test | |
|
fit <- lm(frutos ~ pob, data = mibase) res.fit <- residuals(fit) bartlett.test(res.fit ~ pob, data = mibase) |
Para barlett.test únicamente se puede usar los residuales derivados de una ANOVA (hay algunos trucos para extender su uso, pero finalmente es mejor hacer un gráfico). Un resultado significativo indica que no hay homogeneidad de varianza. |
Sobre colinealidad[editar]
Existe uno problema extra: la colinealidad. La colinealidad surge cuando se agregan variables independientes en un modelo que estan altamente relacionadas entre ellas, son redundantes, o que aportan la misma información (tres definiciones de redundante... ¡qué redundante!). La colinealidad afecta tanto la estimación de los parámetros como su significancia. Aqui presentamos dos métodos para estimar la colinealidad: los coeficientes de correlación (cor) y los valores de inflación de la varianza (vif). La colinealidad estimada a partir de cor solo es examinada entre pares de variables mientras que la estimada por los vif evalúa la información redundante entre una variable y todas las otras. En caso de detectar problemas de colinealidad lo recomendable dejar sólo las variables no redundantes en el análisis y eliminar las otras o utilizar combinaciones de variables que resuman la información (por ejemplo componentes principales).
| cor | |
|---|---|
|
mibase$tam2 <- mibase$tam + rnorm(24, mean = 1, sd = 0.5) cor(mibase[, c("tam", "tam2")], use = "complete.obs") |
Creamos para este ejemplo una variable tam2 que es indéntica a tam mas un pequeño error generado por rnorm.
Valores de correlación por arriba 0.7 indican que las variables son colineales. |
| library(car); vif | |
|
library(car) mibase$tam2 <- mibase$tam + rnorm(24, mean = 1,sd = 0.5) fit <- lm(frutos ~ tam + tam2 + pob, data = mibase) vif(fit) |
Ajustamos un modelo con las tres variables y evaluamos los factores de inflación de la varianza (vif). Si una de las variables fuera un factor con más de dos niveles, R calcula los "vif generalizados". Valores de vif o mayores a 5 indican problemas de colinealidad. |